QIAGEN IPA Summer Release (Jul. 2021夏季更新)

QIAGEN IPA Summer Release (Jul. 2021夏季更新)
QIAGEN IPA 路徑分析軟體在本季度釋出重大更新,提供新模式搜尋及數據分析,詳細更新內容請參閱下方資訊:(若尚未有IPA軟體使用帳號,也歡迎向各單位負責人申請)
A. QIAGEN IPA New Pattern Search! QIAGEN OmicSoft Analyses!
IPA此次釋出新的模式搜索(Pattern Search)功能,可以從可以從網路或生物路徑中的任何基因作為”查詢”起始點,使您能夠立即從>96,000個OmicSoft的資料集進行分析。在使用模式搜索時,可以探討以下幾點問題。
1. 哪些分析與我的資料中的生物路徑擁有相同的基因?
Which analyses activate this Canonical Pathway through the same genes as in my analysis?
2. 哪些患病組織的分析與我的藥物基因特徵“相反”,並且可能受益於哪些的藥物治療?
Which analyses of diseased tissue are the “opposite” of the gene signature for my drug and may benefit from treatment with the drug?
3. 是否有任何分析與我的資料分析中有類似的分子交互網絡?
Do any analyses have an interaction network or Regulator Effects network similar to the one in my analysis?
以下的各式範例圖及實作,你可以從Core Analysis中,選取任一的分子網絡或是生物路徑,或是將感興趣的基因放入”我的路徑”(New My Pathway)”,透過Overlay -> Dataset or MAP,賦予各基因活化或抑制的表現。接著在點選“模式搜索”按鈕,即可發現哪些 OmicSoft 分析具有相似或不同的基因表達模式。進一步也可以創建基因熱圖(heat map),以分析各個基因是如何匹配您感興趣的基因。
例如,下方挑選一組細胞中某些類別的藥物或化學物質,為有轉錄效應的基因特徵(PMID 30552330)。圖1顯示了 My Pathway,其中添加了文獻中的 HDAC 抑製劑。模式搜索將添加的一組上調控和下調控基因,並與 >96,000 個 OmicSoft 數據集中的顯著差異表達基因進行比較。
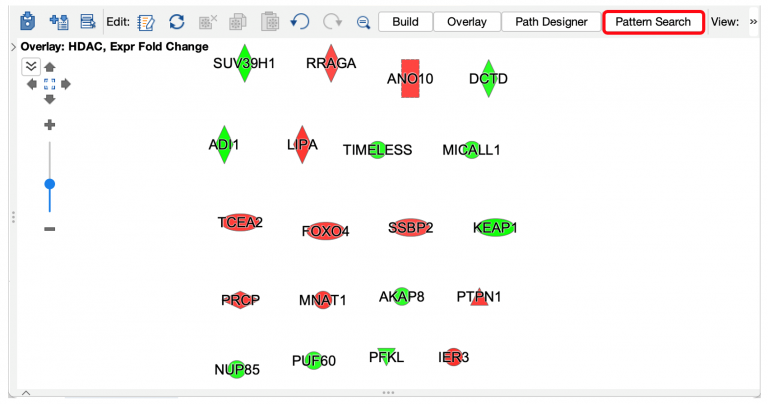
圖 1:My Pathway 中的基因模式(作為查詢起始點)。該特徵代表了 HDAC 抑製劑在細胞中的一致轉錄效應,作者使用 LINCS 數據 ( PMID 30552330 )推導出該效應。畫面上以紅色基因表示會被 HDAC 抑製劑活化,而綠色表示的基因會被抑制。點選紅框顯示的“模式搜索”按鈕,可於 OmicSoft 分析中搜索具有活化抑制匹配或反匹配模式的基因,如圖 2 所示。
- 查詢模式得到許多與癌細胞相關的 LINCS 數據,這些數據已經比對過各種 HDAC 抑製劑的表現,例如 belinostat 和 mocetinostat(見圖 2)。這種數據可用於陽性對照(positive control),因為此文獻室使用LINCS的數據作為資料的判斷。該結果用透過z-score 算法作有效數據集的匹配。
- 此 z-score 算法僅評估每個 OmicSoft 分析中的“分析就緒(analysis-ready)”基因,是否與模式搜尋的查詢匹配。換句話說,在查詢和分析中被活化/上調(紅色)或被抑制/下調(綠色)的基因對 z 分數有顯著的貢獻。
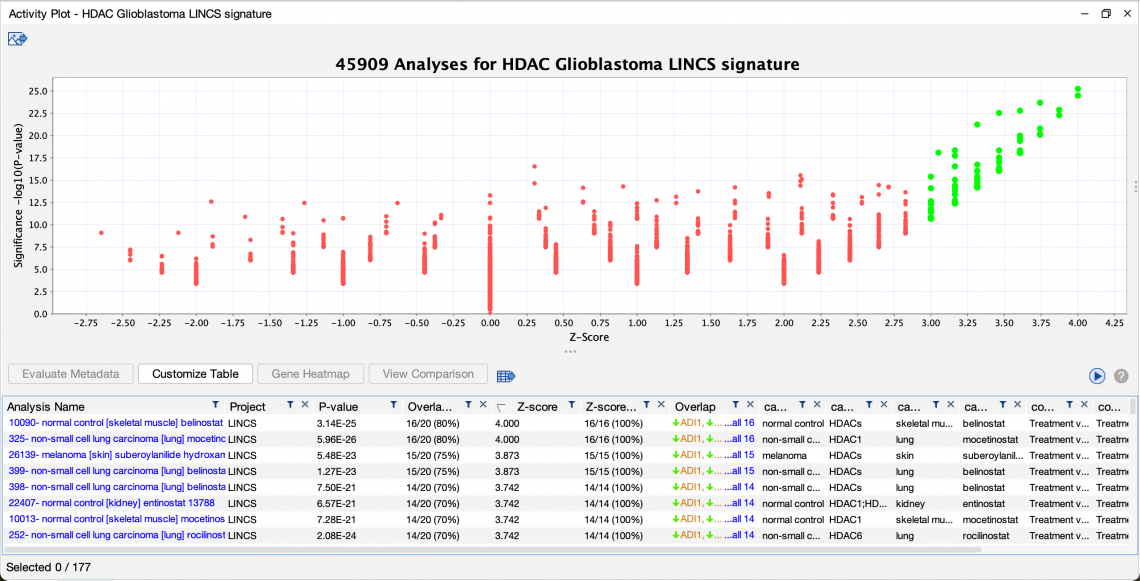
圖 2: HDAC基因特徵與 LINCS HDAC 抑製劑治療研究之各種細胞表現匹配數據。正如預期,模式搜索算法找出與該查詢預期的各種 LINCS 數據集匹配。也可以透過條件篩選LINCS 匹配出的發現(參見圖 3)。
- 我們可以從LINCS 數據集,以各式的條件篩選,檢測到與其他涉及 HDAC 抑製劑治療的實驗匹配,例如神經內分泌腫瘤 (neuroendocrine tumor)、結腸癌 (colon cancer)和膀胱癌 (bladder cancer)的 SAHA 治療(見圖 3)。也可從OmicSoft資料庫比對出與 HDAC 抑製劑無關的數據集。
- 例如,其中有使用Ixazomib治療並作為非耐藥性的對照組細胞株,Ixazomib 為一種蛋白酶體 20S 亞基 beta 5 抑製劑,已被證明與 HDAC 抑製劑在Hodgkin和 T 細胞淋巴瘤中具有協同作用 ( PMID 31452195 )。因此,模式搜索結果發現了許多出乎意料且與生物學相關的數據集。
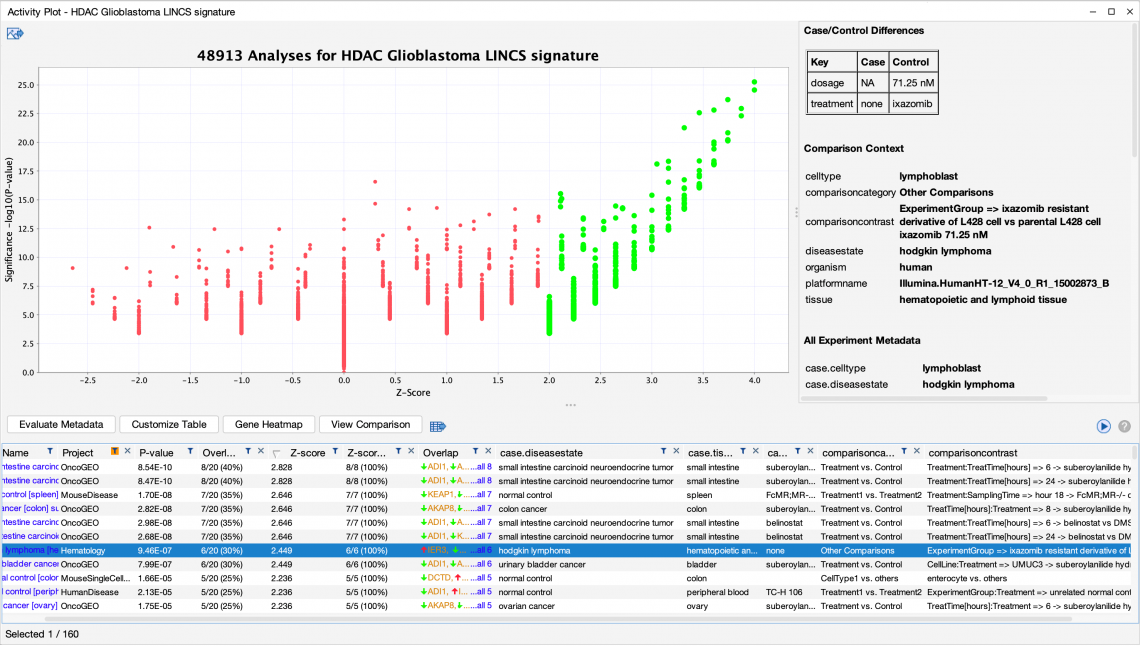
圖 3: HDAC表現特徵也與非 LINCS 數據集具高度匹配。透過篩選圖2中使用的資料來源 (Project),找出了可能額外發現的意外結果。在本例中,HDAC 處理的基因表現與其他治療方式(ixazomib)的分析相匹配。
- 從 Activity Plot(如圖2或3中),您可以輕鬆創建有興趣比較的基因熱圖,以檢查哪些基因在匹配或反匹配分析中,有顯著的基因差異表達。圖 4 顯示了一個基因熱圖,最左邊的列中有查詢分析,其他列則選了其他匹配的 HDAC 抑製劑治療分析。
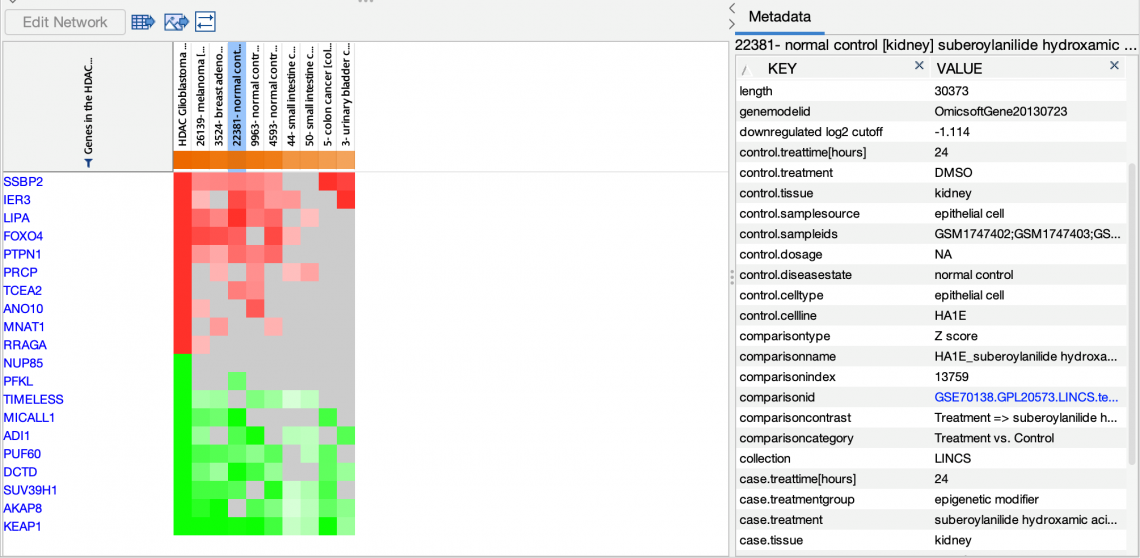
圖 4:利用”基因熱圖”顯示了與所選分析的詳細基因表現變化。在這個例子中,HDAC 特徵在最左邊的列中顯示為紅色或綠色實心方塊,分別表示在匹配分析中預計會被上調控或下調控的基因。顏色強度通過來自 OmicSoft 的一些匹配分析指示實際對數倍數變化。根據用SAHA處理的各種細胞株的篩選分析。所選定的分析以藍色顯示,在右側的表格中顯示其關聯的原始數據。請注意,並不會有任何一個匹配分析與查詢完美匹配,但資料間會的表現基因之間會存在明顯的相似性。
- 值得注意的是,模式搜索適用於您所要查詢網路有上調和下調基因以及透過分子活動預測器 (MAP) 所產生的節點。例如,您可以使用 “Regulator Effects Network” (Core analysis所產生)作為查詢,它顯示您數據集中上調控和下調控的目標基因(分別用紅色和綠色表示),也會顯示預測的上游調控的活化(橘色)和抑制(藍色)。在這種情況下,在”查詢模式”中,紅色顯示為被激活的上游調控,而綠色顯示為被抑制的上游調控。
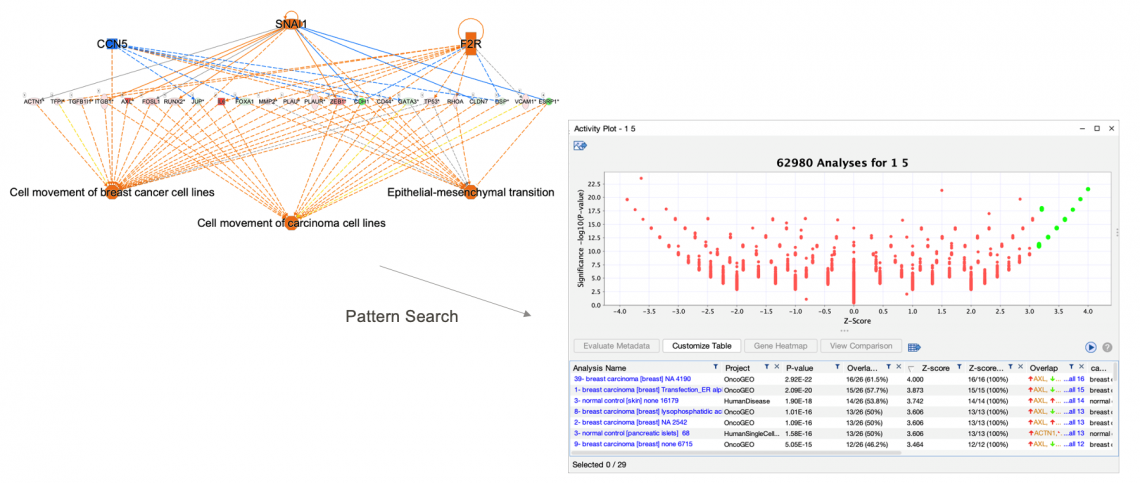
圖 5:模式搜索在查詢中同時使用已知活動和推斷活動。 在此例中,查詢使用 Regulator Effects Network作為查詢。為了搜尋出相關的模式資料,上游調控分子的SNAI1 和 F2R 被視為上調控,CCN5 被視為抑制。
- 模式搜索是透過使用 OmicSoft 數據表現分析,來加強 IPA中生物學解釋的不同方法中的最新方法。圖 6 顯示了每種方法背後的基本原理。
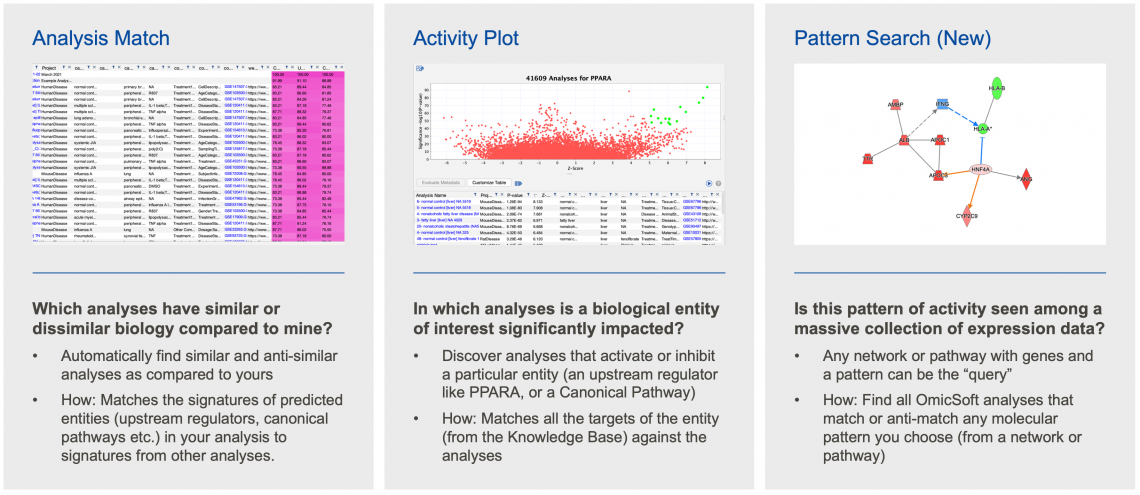
圖 6:IPA 中“分析匹配”功能的三個不同模組。
請注意,模式搜索和圖 6 中顯示的其他功能需要您的 IPA 的具有Analysis Match許可。如果此許可目前無法使用的話,請聯繫使用單位或是授權商創源生技以獲取有關獲得此功能訪問權限的更多資料。
B. Comparison Analysis from Activity Plot results
您現在可以直接從任何活動圖去建立比較分析,如圖 7 所示。
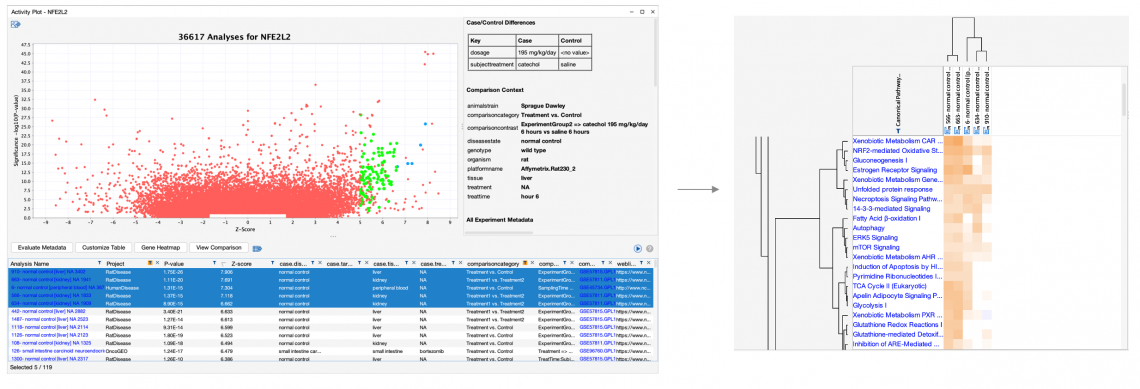
圖 7:直接從ActivitiyPlot活動圖中的選定資料並創建比較分析。在這個例子中,選用上游調控因子NFE2L2之活動圖(左),並用搜索匹配所產生的 OmicSoft Lands 數據集。進一步篩選“比較類別(comparisoncategory)”中僅處理過的樣本中。 在活動圖中選擇前五個結果並點選“查看比較(View Comparison)”後,可以使用生物路徑分析及階層式分層來分析(右)。
C. Newly revised Land Explorer links in Gene Views improve navigation
IPA Gene Views 中的 Land Explorer 資料庫連結,現在提供其他 Lands 和視覺化的直接連結,並且將數據來源的標記更加清楚。今年度的更新,已新增可以鏈接到存活率圖(survival plots)和ICGC(國際癌症基因組聯盟)等來源(圖 8)。
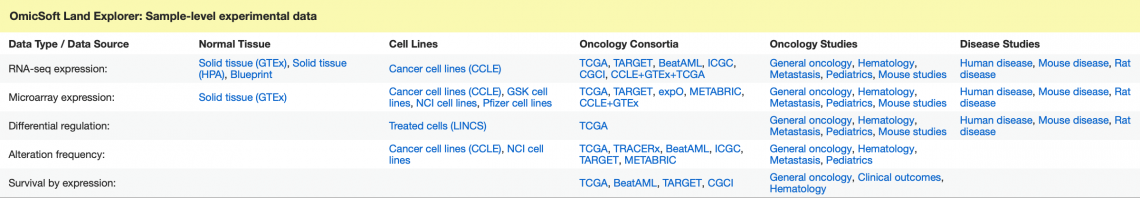
圖 8:基因視圖中優化的 Land Explorer 超連結部分。各連結依照來源、類型、聯盟等分類。
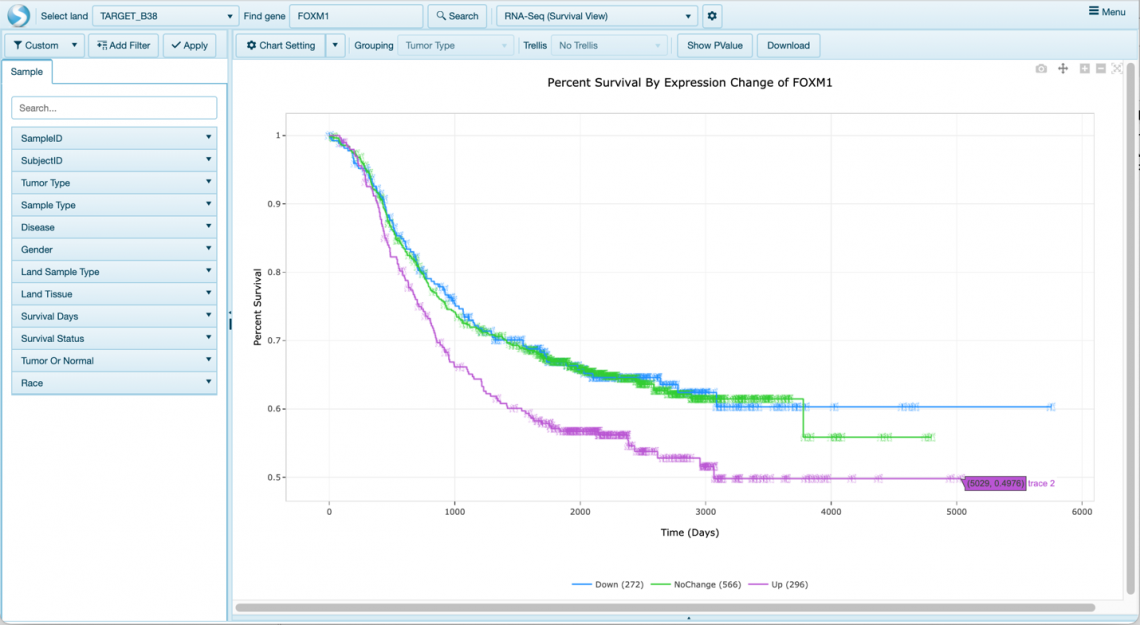
圖 9:Kaplan-Meier 存活率曲線現在可直接連結至IPA中的基因視圖。 該存活率曲線是透過從 FOXM1 Gene View中,依圖 8 所示的 -Oncology Consortia - Survival by Expression - TARGET 生成的。該視圖顯示了隨時間的存活率,並根據該表現進行分類基因。
D. Improve publications by customizing font size in networks and pathways
為了傳達各網路或生物路徑中某些節點或標籤的重要性,可以選擇性地修改字體大小。通過選擇單一、多個節點或標籤,並使用新的字體大小按鈕,即可輕鬆進行修改,如下圖 10 所示。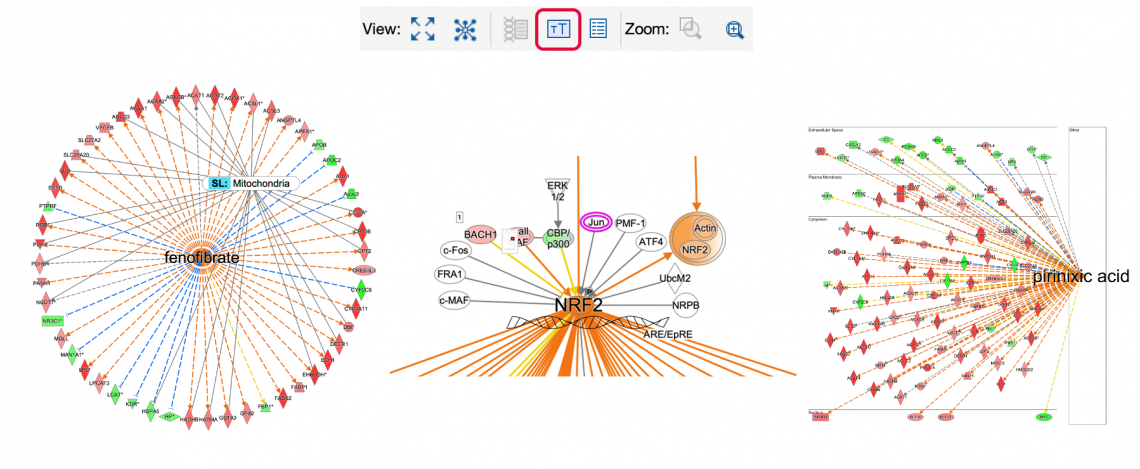
圖 10:現在您可以使用新的字體大小按鈕修改節點或標籤上的字體。這些節點或標籤的重要性可以通過字體大小來強調,並提供與您的路徑圖像在比較上有更好的變化。
E. Content updates 2020 Q3 最新更新Pathway及資料庫數據如下
新增生物路徑:
- CSDE1 Signaling Pathway
更新生物路徑:
- Base Excision Repair Pathway
- Epithelial Adherens Junction Signaling Pathway
- Gustation Pathway
- Phagosome Formation Pathway
並新增OmicSoft資料庫內容如下:
>6000更新資料(總共 > 96,000),並可運用於Analysis Match, Activity Plot and Pattern Search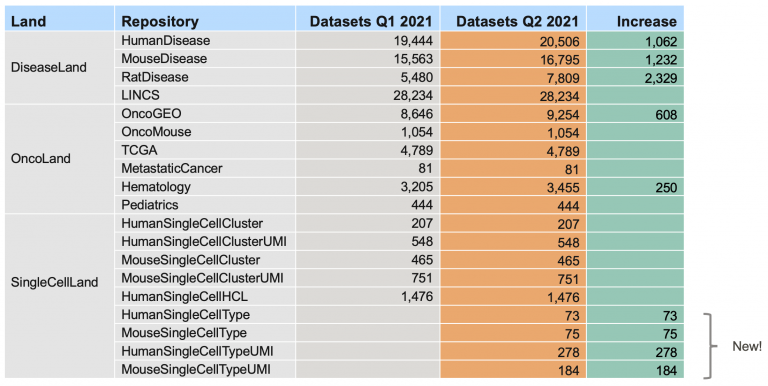
表一、 OmicSoft資料庫更新統計(Analysis Match & Activity Plot)。 超過6,000全新分析數據、1條生物路徑、300,000新增文獻資料。



