QIAGEN CLC – COVID-19 分析範例報告
CLC bio – COVID-19 分析範例報告
一、目的
2019年底至2020年初驚撼人類的新型冠狀病毒(COVID-19),科學家們至今日以繼夜地從科學上的蛛絲馬跡找尋相關藥物,然而新型冠狀病毒的突變種眾多,越來越多研究必須從序列底層著手,才能了解日後的對策,本次CLC可以透過一系列分析流程比對原始參考病毒序列的變異,以及提供組裝指南。
二、材料與方法
(一) 方法
採CLC Genomic Workbench至2020年更新到20版本後,CLC也針對長片段序列平台新增分析模組─Long Reads Support,此模組新增Oxford Nanopore的匯入途徑,以及Nanopore與PacBio的長片段定序資料的拼裝與參考序列組比對功能,更能把長片段組裝後的組裝(contig)資料與短片段片段資訊進行混和拼裝(Polish reads),互補長與短片段在序列拼裝上的障礙性。以下是CLC會使用於變異分析的工作流程圖(Figure 2.1)。
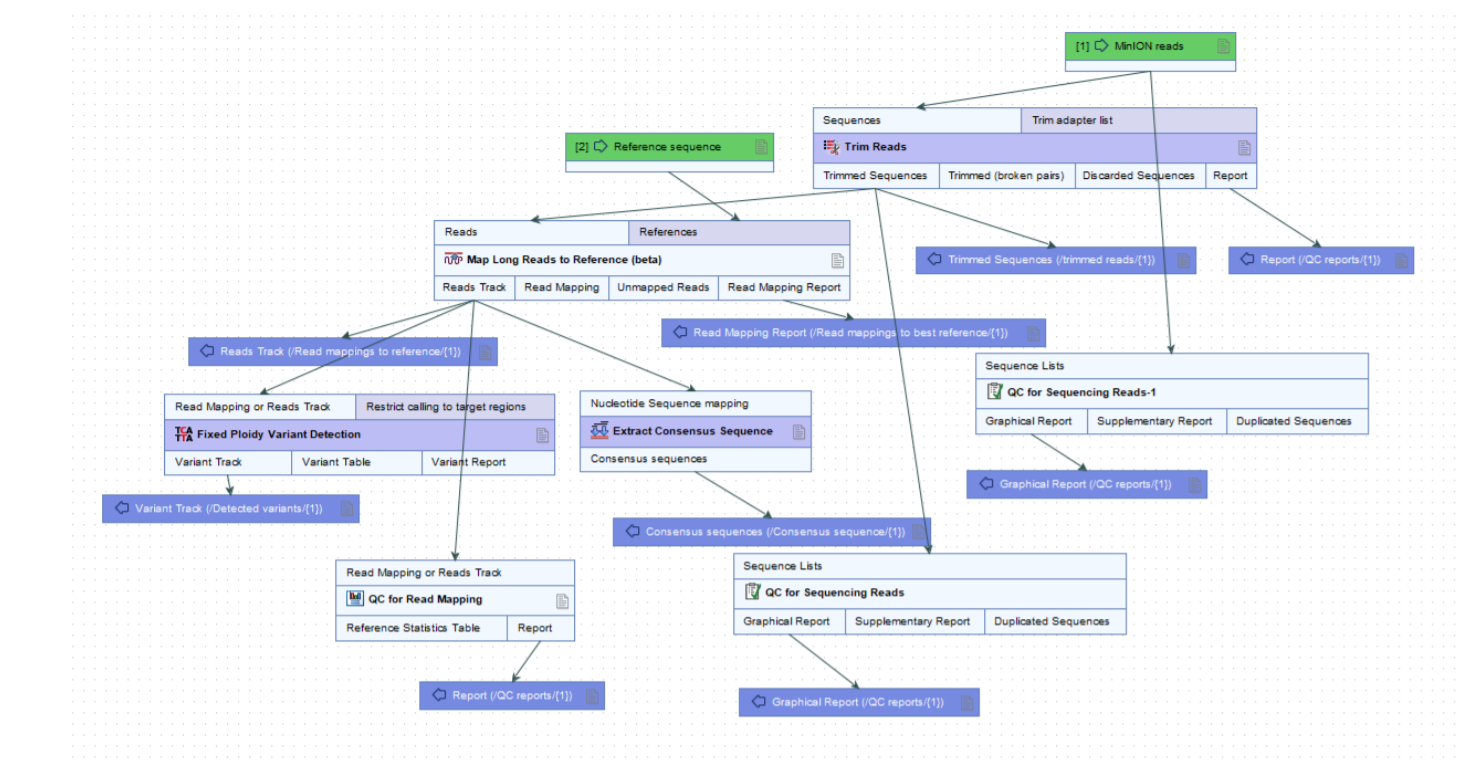
Figure 2.1 CLC變異分析(Long Reads)
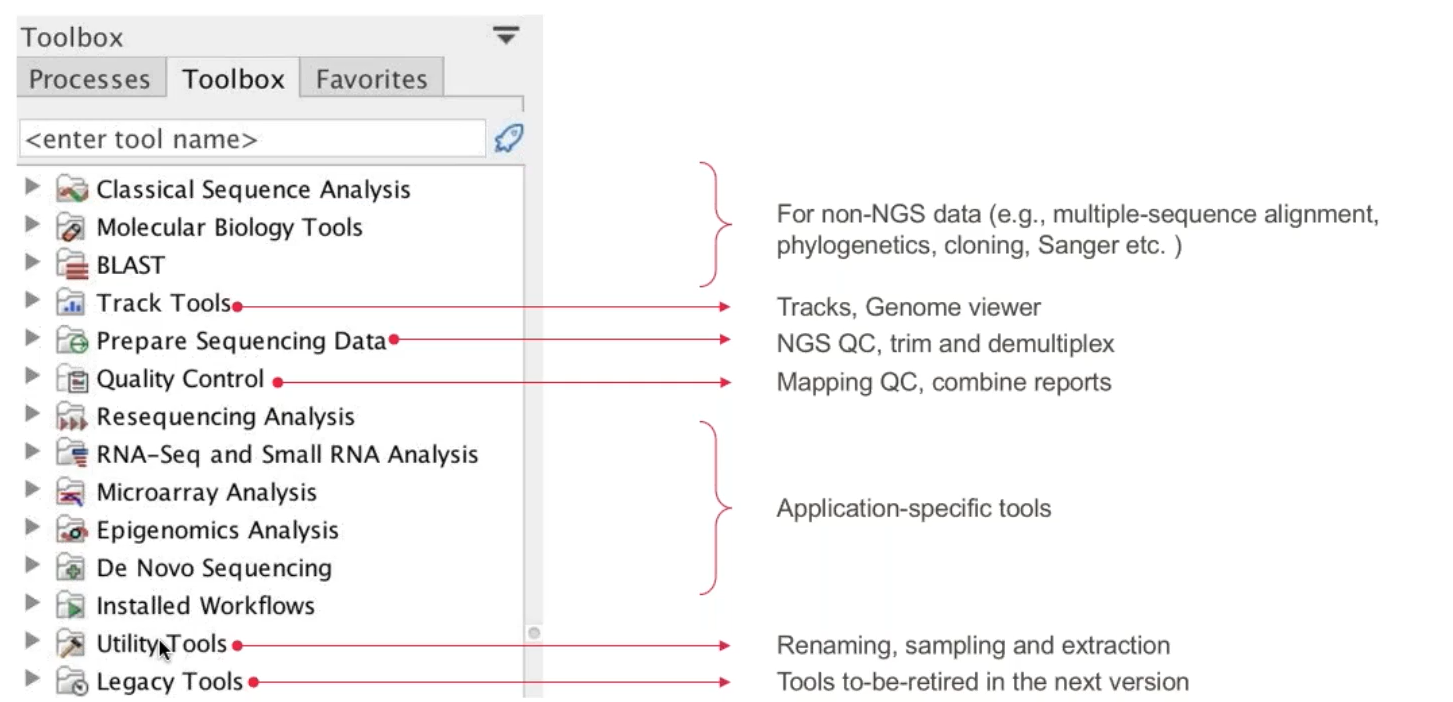
Figure 2.2 CLC基本模組常用功能架構
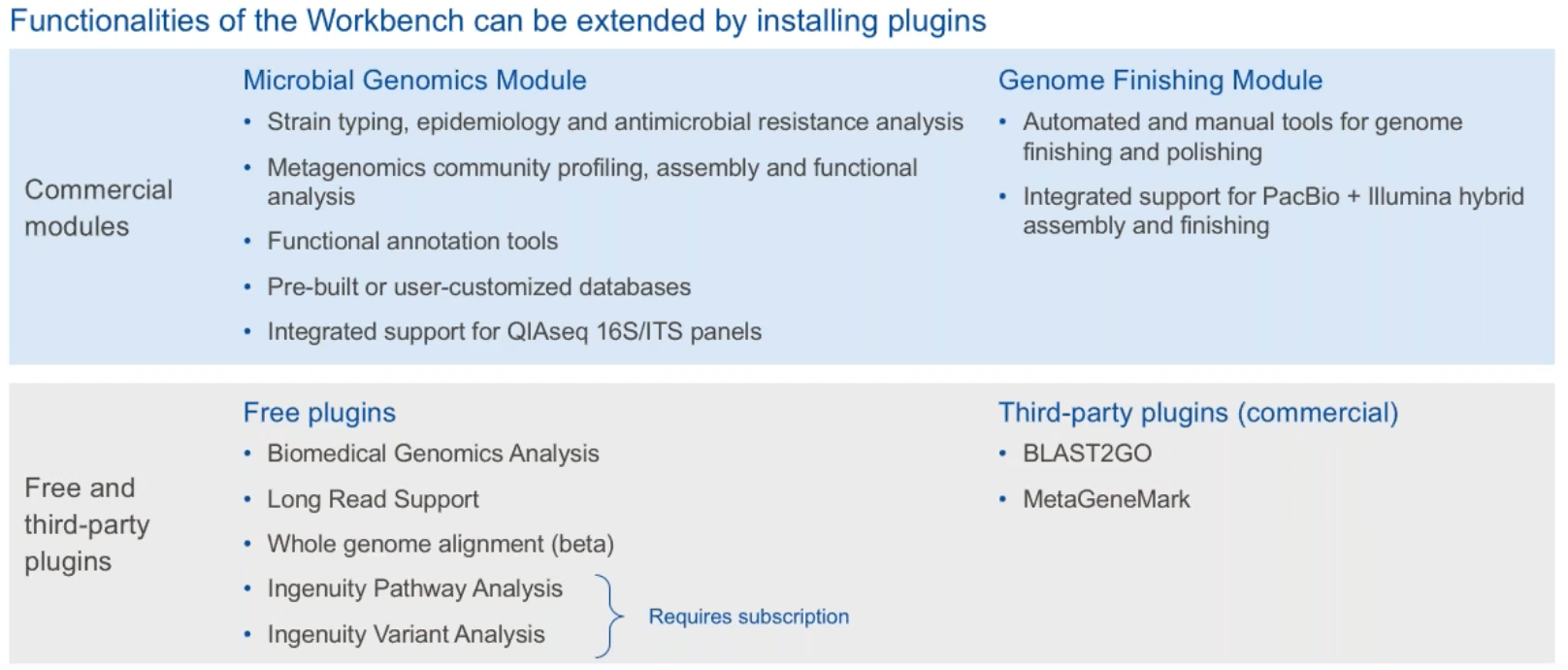
Figure 2.3 CLC擴增模組架構
(二) 材料
1. 2個來自Oxford Nanopore MinION平台的長片段序列資料與SISPA adapter序列
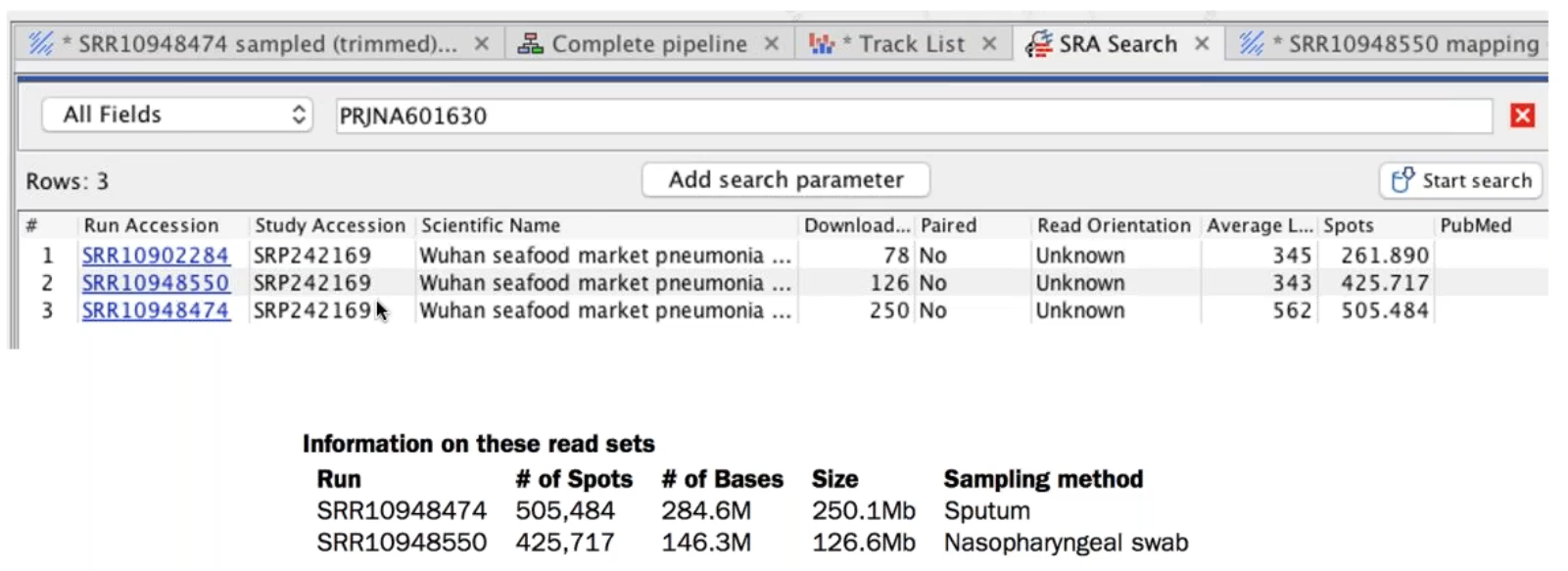
Figure 2.4 CLC內建SRA資料庫下載系統
2. 參考資料庫
SARS-CoV-2序列編號MT135044做為參考基因體
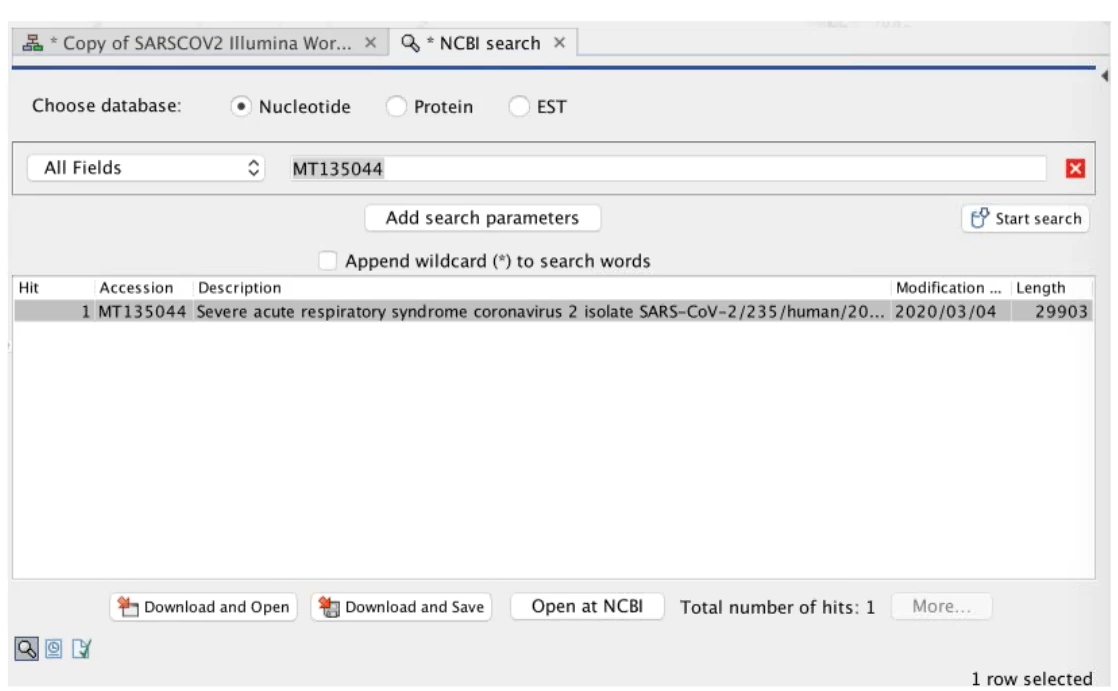
Figure 2.5 MT135044 SARS-CoV-2病毒序列下載
三、變異分析CLC分析產出結果
本次分析結果可分為報告部分(部分顯示)以及圖表部分。
(一) 資料處理部分
為資料前處理,如處理過Quality Control以及Trim Reads所得到的相對應的報告表,反應定序的品質好壞。
1. QC Report:
在此顯示報告內Quality distribution,讓使用者了解資料內的品質程度(Figure 3.1)。報表除了簡易QC的Summary,還包含:Length distribution、GC content、Ambiguous base-content、Quality distribution,以及與Coverage有關的資料。
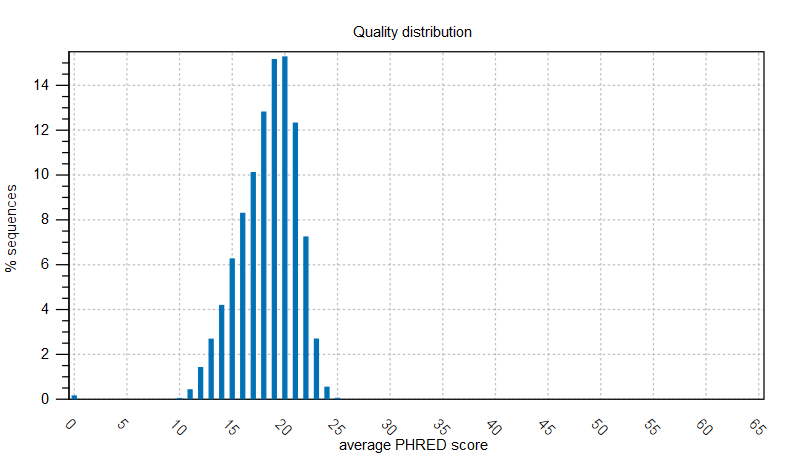
Figure 3.1 Quality distribution
2. Trim Reads:對於每一條序列上品質較不好的區域進行修飾的過程。報表包含Summary報表、 各個樣本在Trim reads上的簡述,以及Trim前後的distribution圖示(Figure 3.2)。
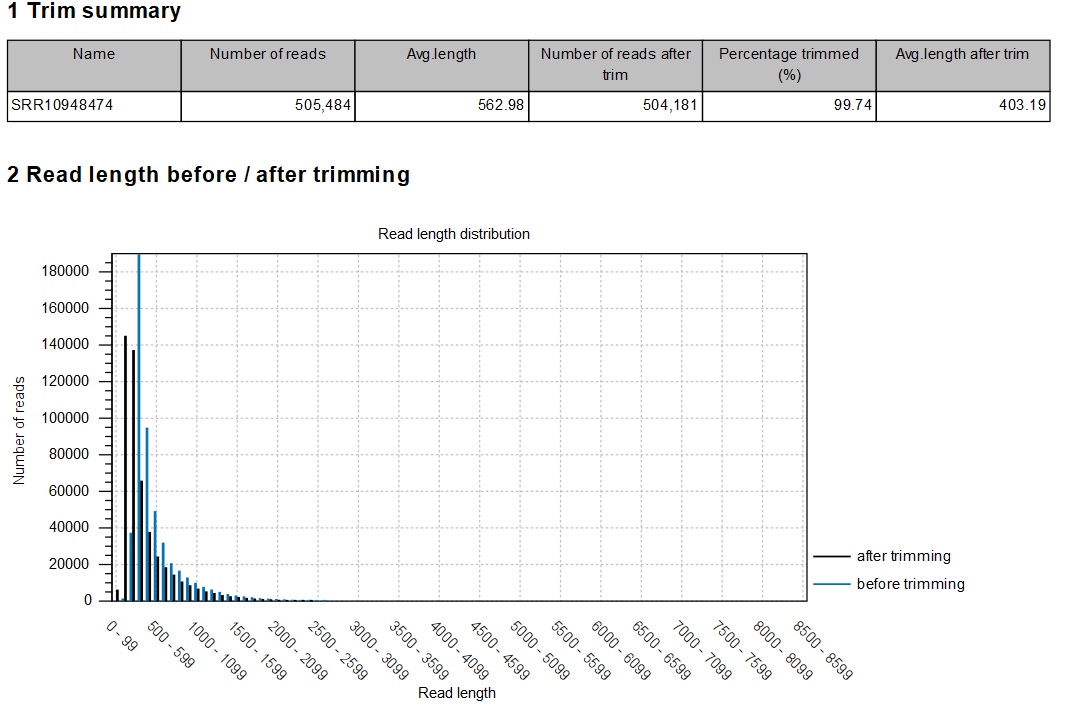
Figure 3.2 Trim reads後的報表形式(部分)
3. Mapping reads report (Table 3.1)。
從Mapping報告可以知道整體序列資料和參考序列新冠病毒序列MT135044在定序上的差異性,包含實際序列比對的對應比率,進而了解定序的品質與評估後續的分析內容。其序列覆蓋率在這次資料中並沒有很好,與早期這資料在病毒萃取(isolation)的過程中是有關係的。
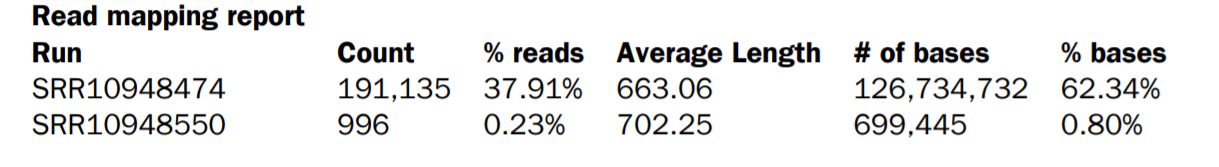
Table 3.1 檢定每個樣本對應到SARS-CoV-2參考序列的序列覆蓋率
(二) 變異分析結果部分
1. 變異點列表
CLC不僅在人體上有預定建置分析流程,透過此分析方式可以找出和原新冠病毒不同的變異位點,從本次例子來說,兩個樣本在比對的情況下找到三個類似且出現的變異位點(分別位於4402、5062與29095位置上)。額外CLC也提供位點比對功能,可以分別針對配對組織、分群或組內進行位點的差異性比對,來找出一群具備特異性的變異點組合。

Table 3.2 變異點分析報表(部分顯示)
2. 基因瀏覽器
實際上可以將長片段nanopore序列資料的比對檔案(.bam格式)與抓到變異點資料實際上在CLC的基因體瀏覽器下觀察變異點的實際狀況,來確保真實變異點的差是否有來自因為演算法的差異,或者是長片段資料帶來的定序偏差效應。CLC的基因體瀏覽器可以在變異點清單下挑選變異點的時候,自動產生變異點在基因體上的資訊,同時也能萃取該變異點的序列片段(非該原始序列)。
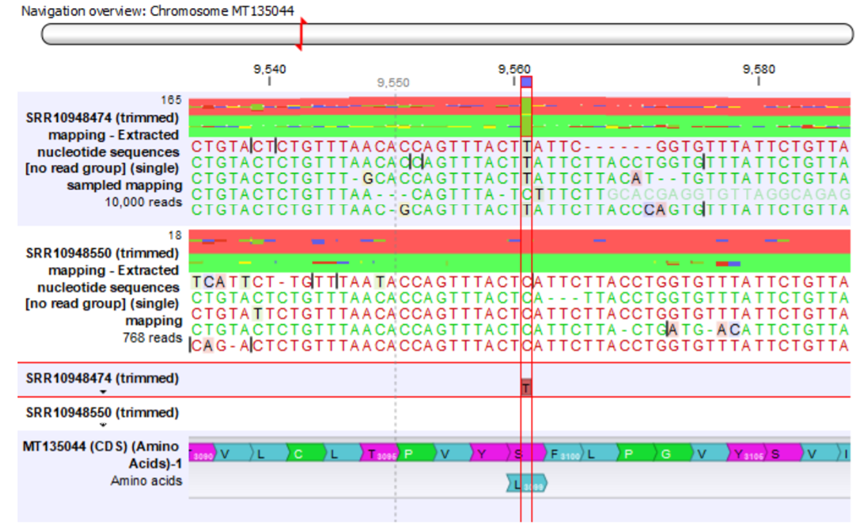
Figure 3.3 變異點瀏覽與基因瀏覽器
3. 萃取出一致性序列
變異點的序列可以從「Extract consensus sequence」的功能抓出和原始貼附序列的一致性片段(consensus sequence),搭配CLC的BLAST功能,從NCBI或自建序列資料內比對序列相似度,可以進行後續註解或找尋演化差異性的研究。如果使用者額外有使用BLAST2GO的軟體,CLC有與BLAST2GO支援模組,可以將分析結果直接遠端連線到BLAST2GO分析,而其分析結果同樣也會部份回傳回CLC軟體內呈現。
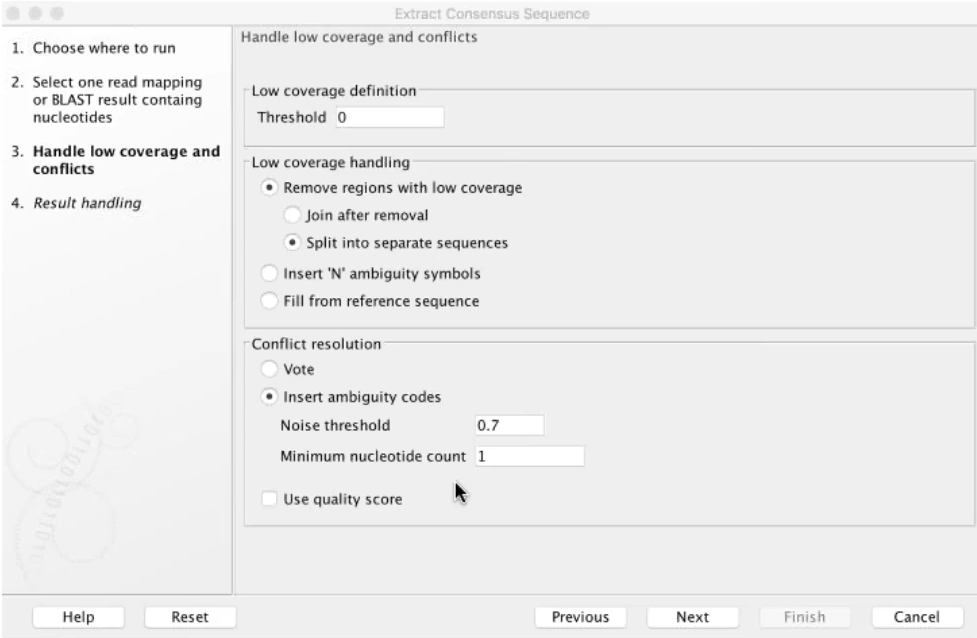
Figure 3.4 獲取一致性序列設定
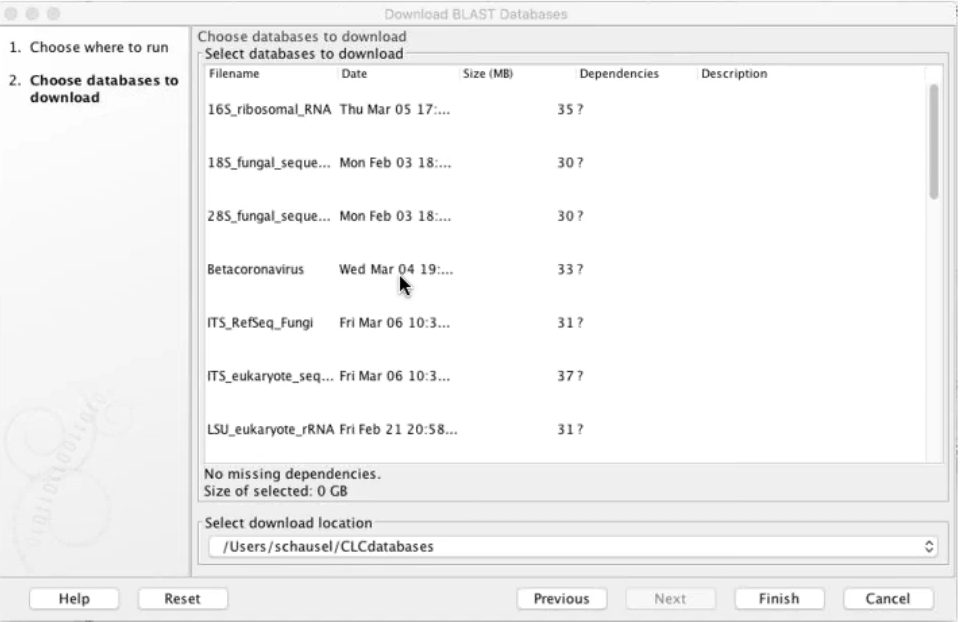
Figure 3.5 可以內建微生物或病毒序列的BLAST資料庫
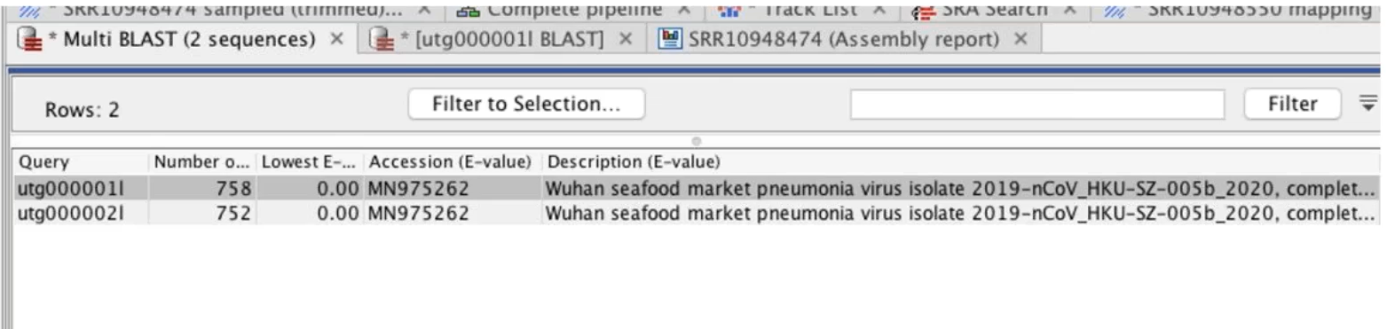
Figure 3.6 將SARS-CoV-2的一致性序列比對於Corona virus的BLAST資料庫
四、基因體序列分析
以下是利用Long Reads Support,長片段組裝後的組裝(contig)資料功能。因此在這裡提供使用CLC的分析流程來分析COVID-19。
(一) De novo assemble long reads
使用者可以調整參數如work size等去將稍長的序列組裝成長片段組裝序列。如果有短片段序列更可以混和其短序列後得到更好的成果。
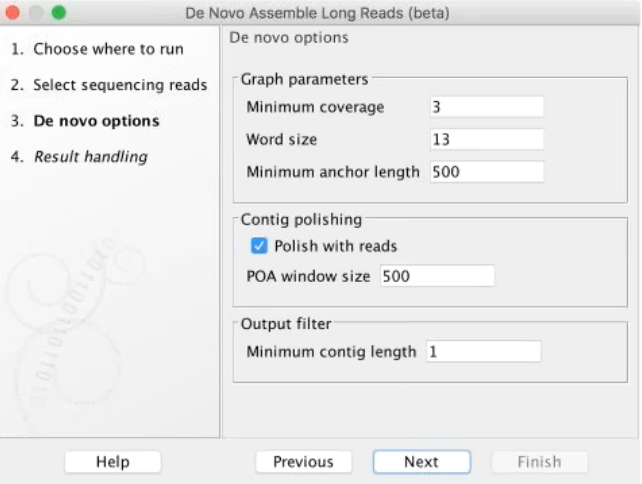
Figure 4.1 長片段組裝的參數設定
(二) Whole genome alignment
CLC有很多不需額外付費的擴增模組,其中Whole genome alignment模組可以將不同長片段的基因體序列比對在一起,來知道彼此之間序列上的差異性。其下圖建立出點比對圖,來知道哪些序列上一致程度的高低。
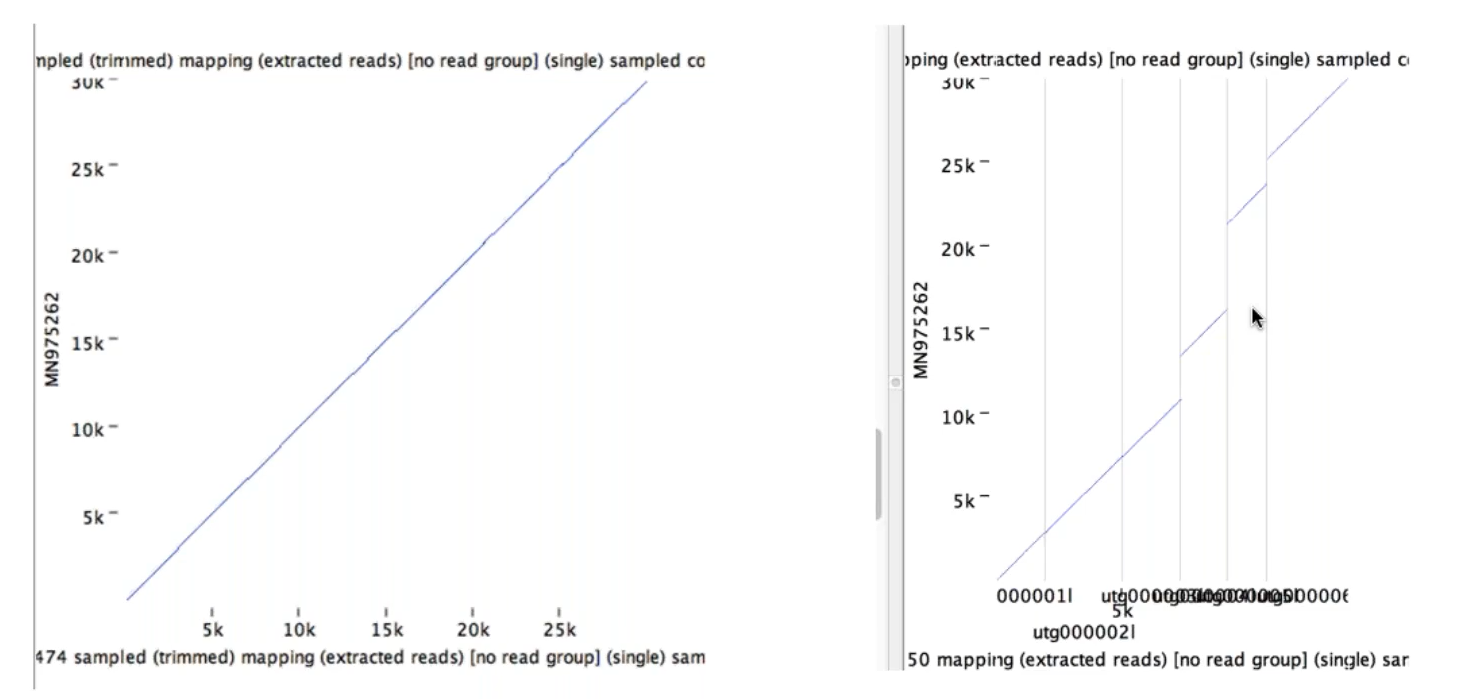
Figure 4.2 點比對圖的呈現可以知道那些序列組裝的一致性程度



