QIAGEN IPA Summer Release - 2023 夏季更新所新增的功能
在 Analysis Match 中使用更精確的配對方法
在最新的版本更新中,我們推出了一種新的配對方法,可以將您上傳的分析資料中可以分析的基因與 IPA 內的 OmicSoft 資料庫中所有的分析資料進行配對分數計算。這與原有的方法 「Analysis Match」有所區隔,新的配對方法專注於基因層面的配對,藉由評估基因之間的相似性來確定上傳的資料與 OmicSoft 資料庫資料間的關聯性。相較於先前的方法,新方法不再只集中在上游調節因子和經典路徑等生物學功能的重疊程度。
這項新方法被命名為「Dataset Matching」,其主要特色在於配對過程主要是在資料集基因的層面上進行的。新的分數出現在 Analysis Match 表中的最右列,相鄰於原有的整體 overall z-score 欄位。新的方法可以比之前的配對方法更精確。不僅如此,它還可以用於配對非常小的資料集, 像是少於 100 個基因甚至只有 10-20 個基因的資料集。儘管新的資料集配對方法更精確,但他提供的分析結果在基因層面相關性較高,而在生物學層面相關性較低。這是因為該方法主要通過比對資料集基因來確定相似性,而忽略了其他生物學功能和關聯。因此,如果您對較高層面的「生物學」相關性感興趣,可能需要考慮使用原有的分析配對方法。
圖 1 描述了在同一個分析的 Analysis Match 結果中,分別以原有分數 (overall z-score) 和新分數 (dataset match) 進行排序的結果。根據 dataset match 分數進行排序 (圖 1 的上圖)得到的結果似乎比原有方法(圖 1 的下圖)更能避免連結到預期外的組織。這項結果凸顯出,採用 dataset match 分數進行排序的方法,能夠得到更符合預期的配對結果。這意味著新的評分方法能夠更準確地找到與目標分析相關的分析結果,可能更符合研究者的預期。
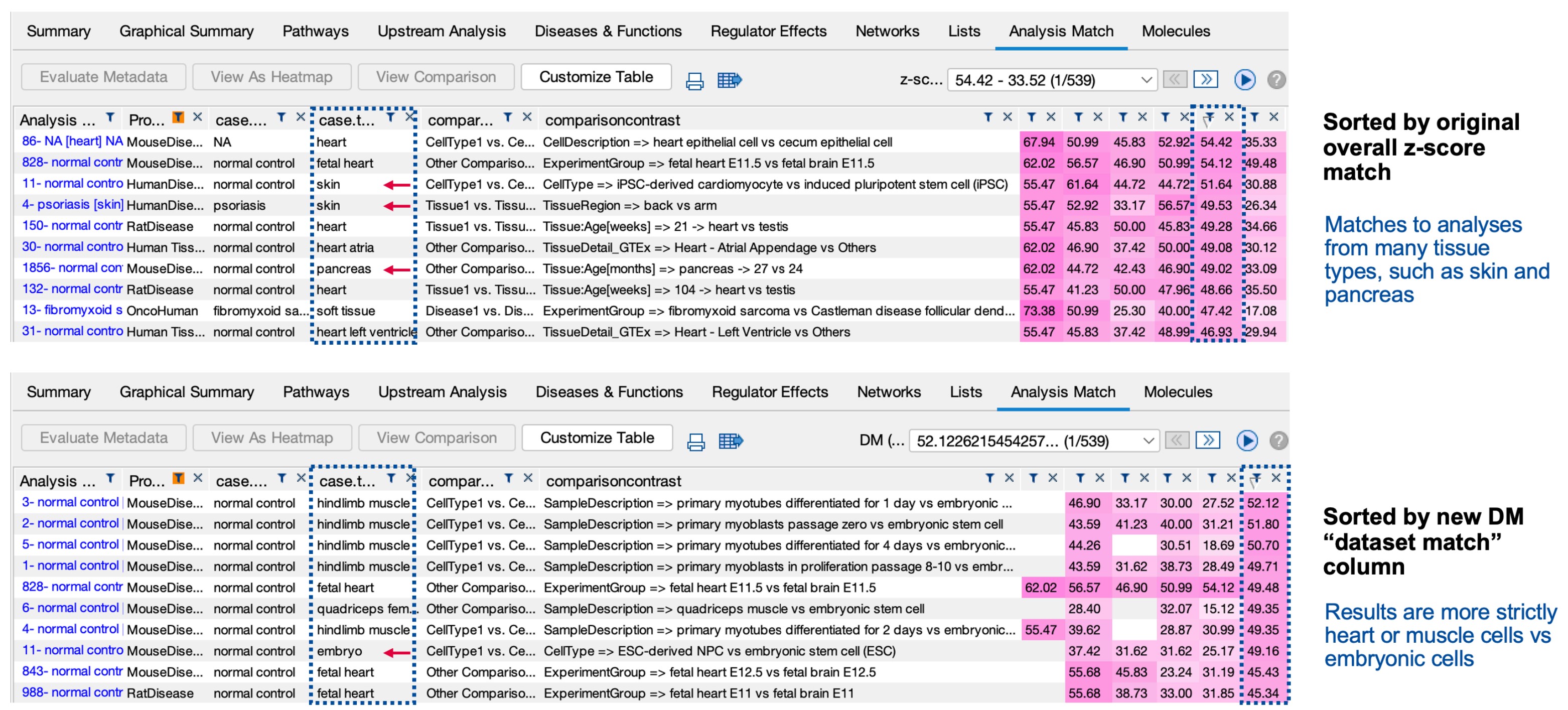
圖 1、根據 overall z-score 得分(上)和 dataset match 得分(下)排序的 Analysis Match 結果。
使用心肌細胞對比胚胎幹細胞的研究進行配對分析,其中紅色箭頭標示了非預期的組織。 下方的表格顯示,新推出的「dataset match」評分方法相較於原有的技術,更能有效減少與 預期不符的組織連結。
在選擇要與您查詢的分析進行比較的分析之後,可以藉由建立一個熱圖來查看查詢分析和配對分析之間的基因重疊集合,如圖 2 所示,然後點擊標示為「Analysis-ready genes」的行中感興趣的熱圖方塊即可。
要更進一步查看您所查詢分析的結果與配對到的分析結果之間的基因重疊集合,您可以依循以下步驟操作:
1. 首先,選擇要與您查詢的分析結果進行比較的分析,以建立一個熱圖。
2. 在建立熱圖後,您可以在標有「Analysis-ready genes」的行中,點擊感興趣的熱圖方塊。
3. 點擊熱圖方塊後,將會打開一個窗口,其中顯示了查詢分析和配對分析之間重疊的基因集合。
透過這項方法,您可以在熱圖中直接觀看查詢分析和配對分析之間重疊的基因集合,以便更詳細地瞭解它們之間的關係和相似性。
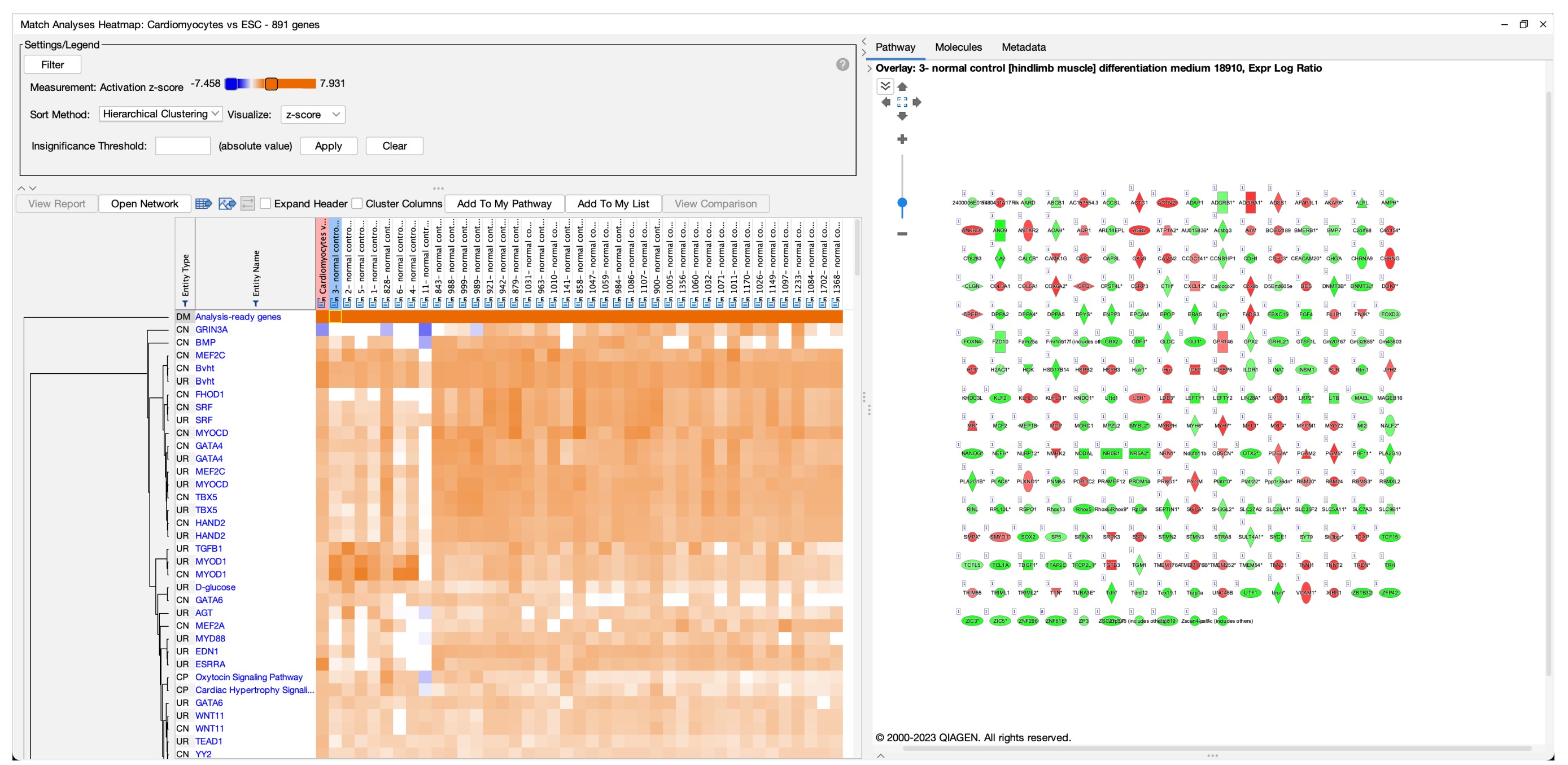
圖 2、前四十名配對分析的熱圖。
熱圖最上面一行表示查詢資料集中的分析就緒基因與所有選擇的分析結果進行配對得到的 z-score。較高的 z-score 反映了兩個分析之間的基因集具有更高的相似性,而較低的 z-score 則表示它們之間的基因集存在較大的差異性。通過比較 z-score,您可以評估分析結果之間的相關性,並挖掘出可能與您的分析結果最緊密相關的其他分析。最左邊的亮橙色方塊是查詢分析中的分析就緒(analysis-ready,AR)基因與查詢資料本身之間的「自我」配對, 該配對在粉色列中顯示。點擊其中一個方塊將在相鄰的窗格中打開一個通往「Sample to Insight」路徑,該窗格顯示查詢分析中的分析就緒基因與配對分析之間的重疊基因(更深 入的細節可見於圖 3)。
 圖 3、心肌細胞分析和其最佳配對分析之間有 250 個基因配對結果。
圖 3、心肌細胞分析和其最佳配對分析之間有 250 個基因配對結果。
以「心肌細胞對比胚胎幹細胞」分析(來自 GSE47948, PMID: 22981692)為例,其結果與一項肌肉纖維束對比胚胎幹細胞的分析結果高度配對(GSE63136, PMID: 25801824)。透過點擊熱圖方塊並使用重疊工具中的「Analyses, Datasets & Lists」功能手動將分析結果覆蓋在上面,建立了這個路徑圖。值得一提的是,這兩個分析中的全部 250 個基因在基因表達趨勢上完全一致,換言之,同一個基因在兩個分析中的表現量皆呈上升或下降趨勢。
新的配對分數計算方法 (Dataset Matching) 通常適用於小型資料集,這些資料集的基因數量通常相對較少,無法生成強大的上游調控因子、因果網路、經典路徑和疾病功能特徵以配對其他分析結果。 以心肌細胞對比胚胎幹細胞分析為例,僅選取前 10 個基因進行匹配分析(按照 p 值和 fold change 值排序)。我們可以觀察到,每個配對分析都有一個獨特的分數,此分數精確顯示了配對分析與心肌細胞對比胚胎幹細胞資料集的匹配程度。分數越高,匹配程度則越精確。
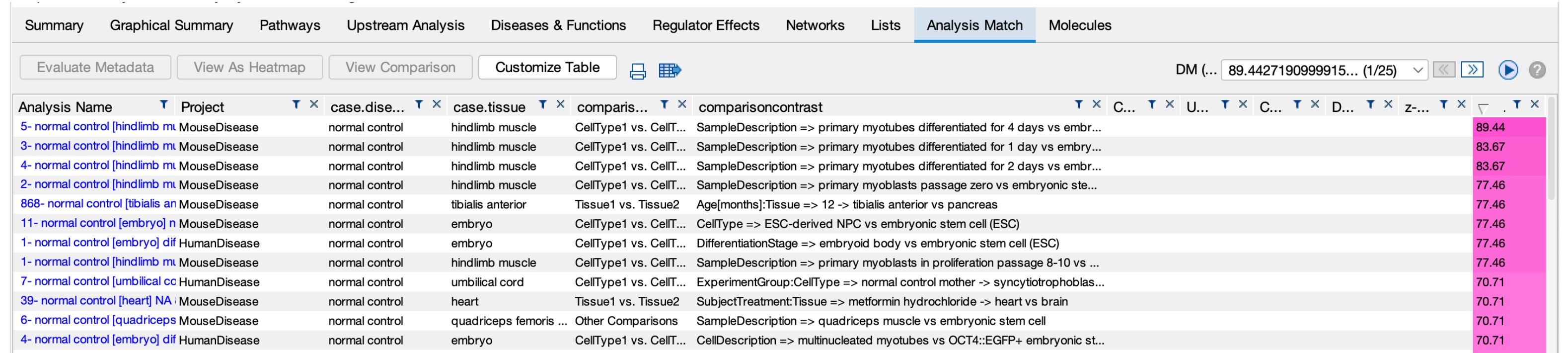
圖 4、使用小型資料集的 Dataset Matching 評分方法。
以心肌細胞對比胚胎幹細胞分析中的前 10 個基因進行配對分析,可以得到與預期類型的分析較配對的結果。這意味著這個新的評分方法在處理小型資料集時能夠提供更準確的結果。
新的資料集配對評分方法與原有的評分方法相輔相成,新的評分方法提供了更精確的配對方 式,使您可以發現更多資料集之間的相關性,進而開拓更多有價值的研究方向。我們期待您能 夠利用這種新的評分方法進行探索,並從中獲得豐富的研究成果!
利用機器學習在核心分析中進行疾病路徑的分析來發現新的關聯性
大約一年前,使用機器學習(ML)技術建立了約 1,500 個疾病和表型網路,並在 IPA 中推出了「ML Disease Pathways」 分析結果(之前稱為「Inferred Networks」)。這些網路包含已知會對疾病路徑造成影響的基因和蛋白質,並提供了機器學習推測的分子,而這些分子尚未被文獻證實參與疾病路徑或尚未被整理到 IPA 的資料庫中。
現在,當您進行核心分析時,這些機器學習網路將根據 z-score 和 p-value 自動對您的資料集進行評分。這些結果提供您的分析資料與疾病之間的潛在新關係。例如,圖 5 展示了機器學習路徑如何評分使用辛伐他汀(simvastatin)處理的人類HUVEC細胞的轉錄組資料(資料來自GSE85799)。
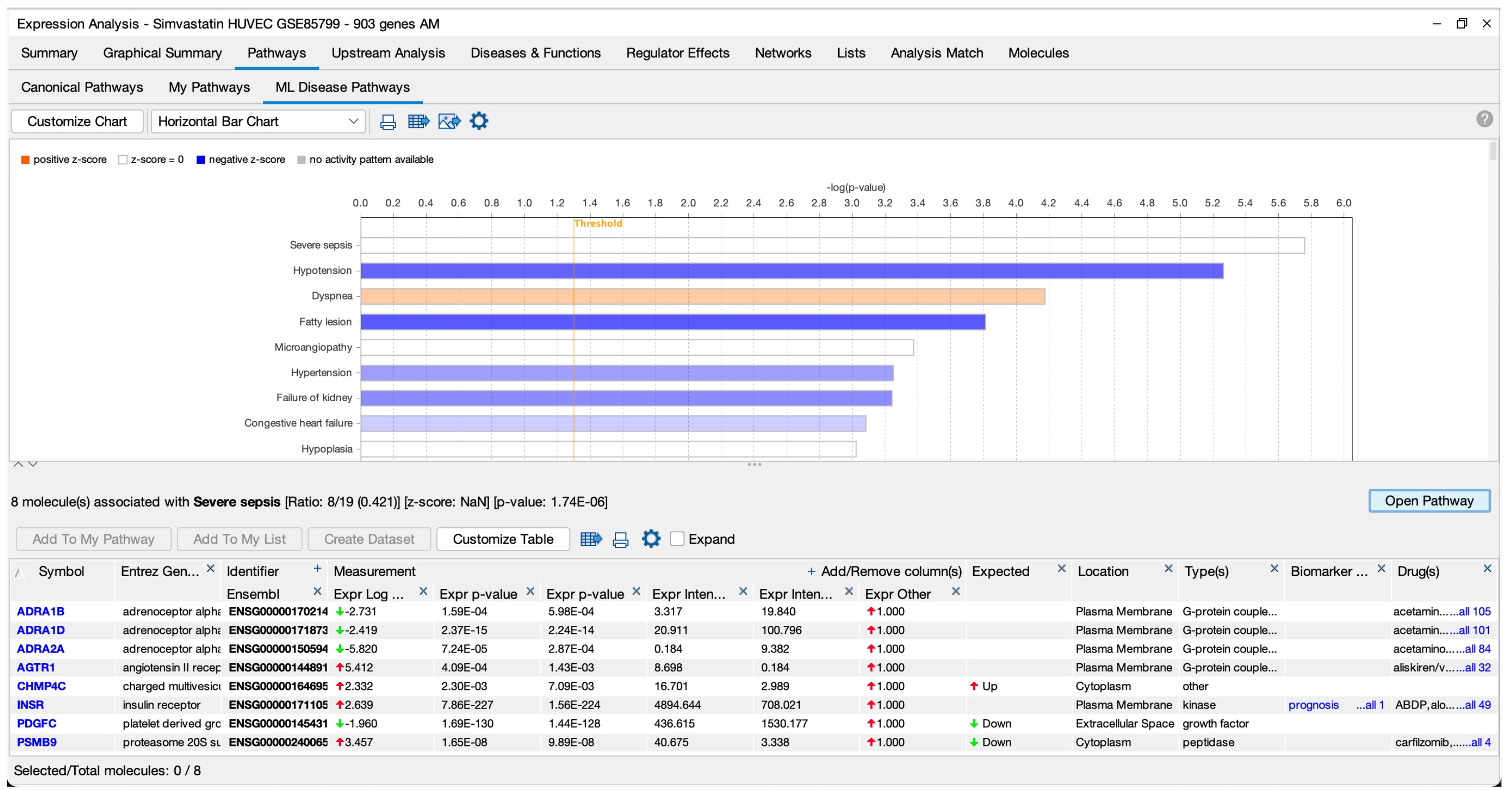
圖 5、對使用辛伐他汀治療的大鼠(肝臟)進行評分的機器學習疾病路徑。
依照 p 值排序,最顯著的結果是「嚴重敗血症」。
點擊「嚴重敗血症」的長條圖會顯示其路徑圖,如圖 6 所示。該圖顯示了使用辛伐他汀治療後 的基因表達模式(紅色或綠色節點)與預測會被影響的相鄰分子(橙色或藍色節點),結果顯示該藥物可能降低敗血症的發生。有趣的是,IPA 中辛伐他汀的 Chem View 頁面顯示該藥物正在進行第 4 期臨床試驗,試驗對象是敗血症(雖然不是嚴重敗血症,但與這個例子相關)。
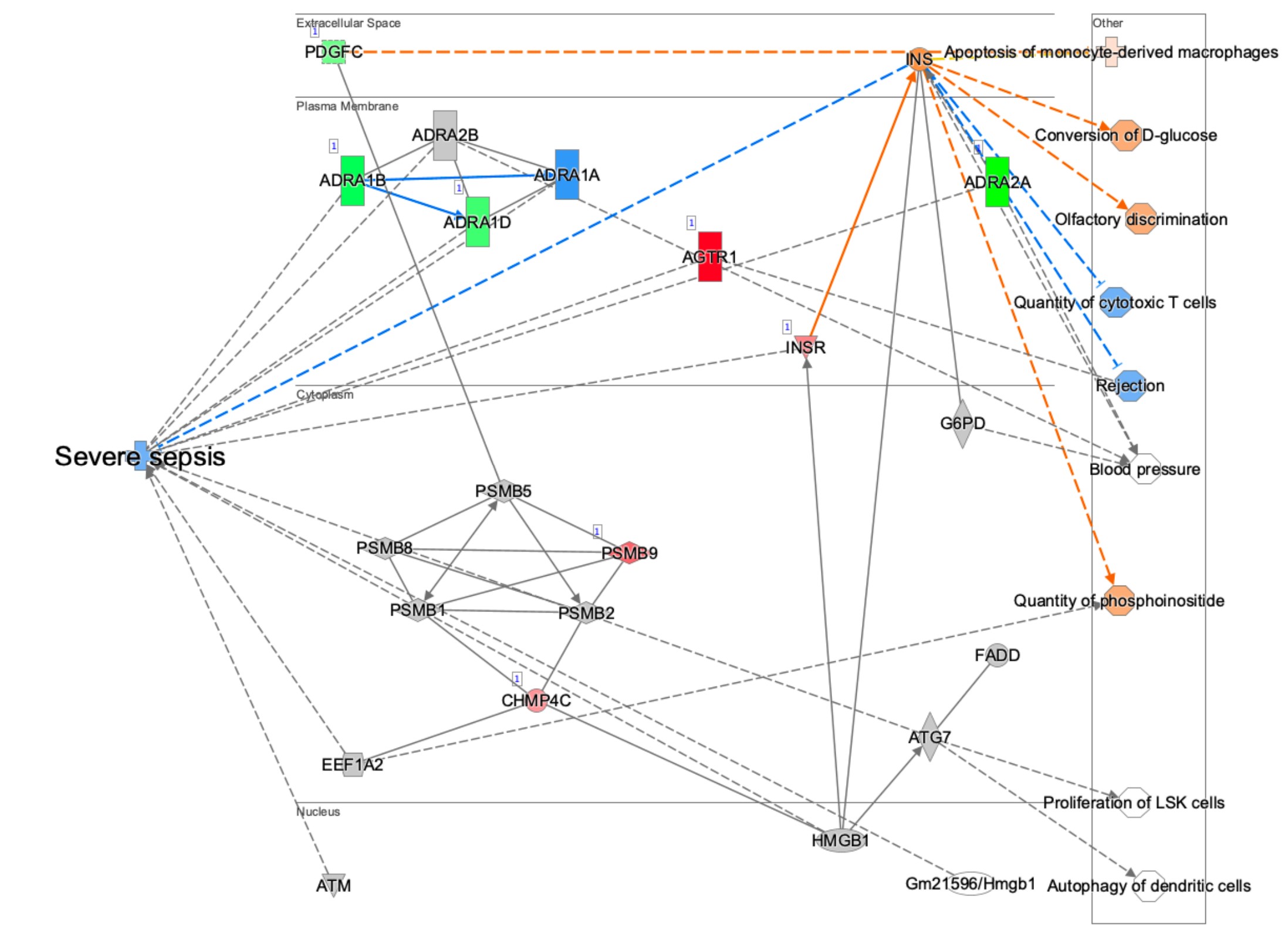
圖 6、嚴重敗血症的機器學習路徑,並與辛伐他汀的差異表達資料進行了疊加。
根據 IPA 的預測,辛伐他汀可能降低嚴重敗血症的發生。
快速且輕鬆地選擇要在圖表中顯示的經典路徑(Canonical Pathways)
為了使您能更方便地在圖表中選擇要顯示的經典路徑,我們在「Customize Chart」方塊中新增了一個自動完成框。如果您希望排除某個特定路徑,只需輸入該路徑名稱的部分文字,然後在搜尋結果中取消勾選即可。另一方面,如果您想快速專注於一個或多個路徑,您可以先取消勾選「Select All」,然後輸入與路徑相關的文字,以找到您想要包含的路徑,最後勾選它們。圖 7 顯示了一個範例,其中使用者只想在圖表中顯示與肌動蛋白相關的路徑。
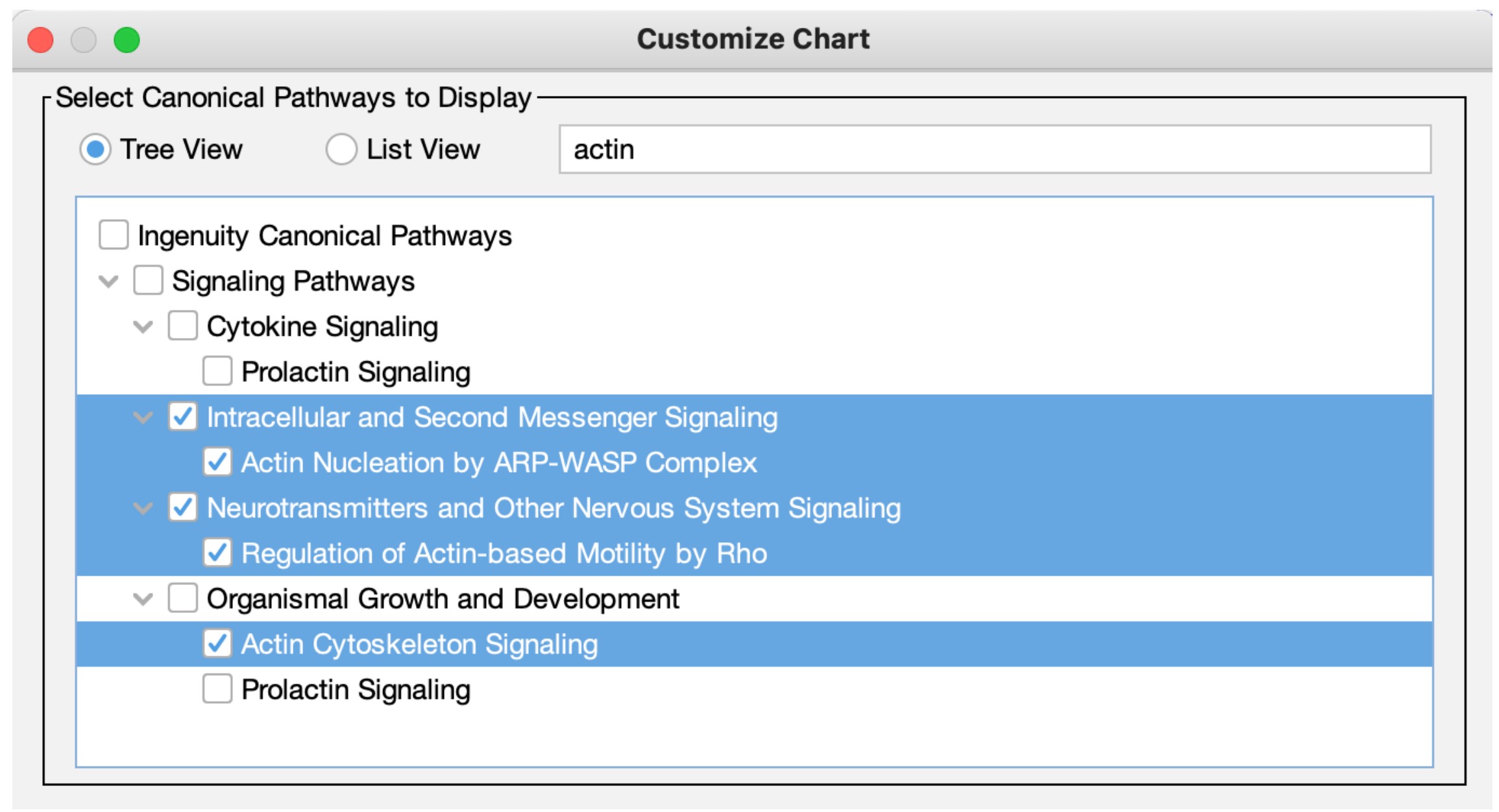
圖 7、在 Customize Chart 方塊中快速專注於感興趣的經典路徑。
資料庫更新
新增生物路徑
• Acetylcholine Receptor Signaling Pathway • Adrenergic Receptor Signaling Pathway (Enhanced)
• Cachexia Signaling Pathway
• GABAergic Receptor Signaling Pathway (Enhanced)
• Glutaminergic Receptor Signaling Pathway (Enhanced)
• ISGylation Signaling Pathway
• Microautophagy Signaling Pathway
• NFKBIE Signaling Pathway
• Orexin Signaling Pathway
• Sertoli Cell Germ Cell Junction Signaling Pathway (Enhanced)
更新生物路徑
• IL-17A Signaling in Fibroblasts
• Sertoli Cell-Sertoli Cell Junction Signaling
• TR/RXR Activation
新增超過 400,000 個新資料(使 IPA 資料庫的資料總數超過 1260 萬筆)
>29,000 protein-protein interaction findings from BioGrid
>407,000 cancer mutation findings from ClinVar
>1,800 target-to-disease findings from ClinicalTrials.gov
>1,700 drug-to-disease findings from ClinicalTrials.gov
>800 Gene -Ontology findings
>220 mappable chemicals
> 3,800 Lipid Maps IDs
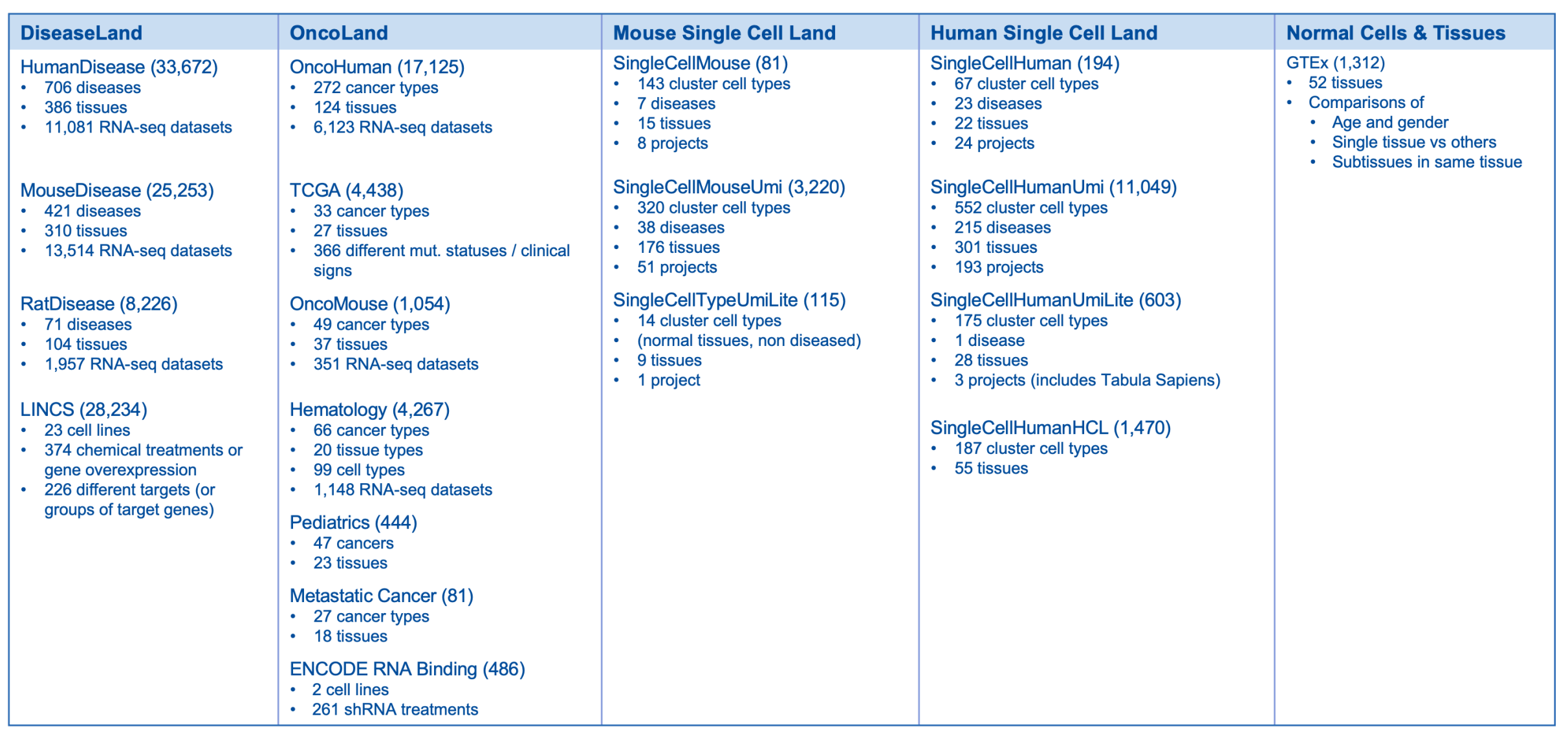
新增 OmicSoft 資料庫內容如下,到 2023 年 7 月,將提供 141,323 個基因表達資料集(新增 5,689 個)
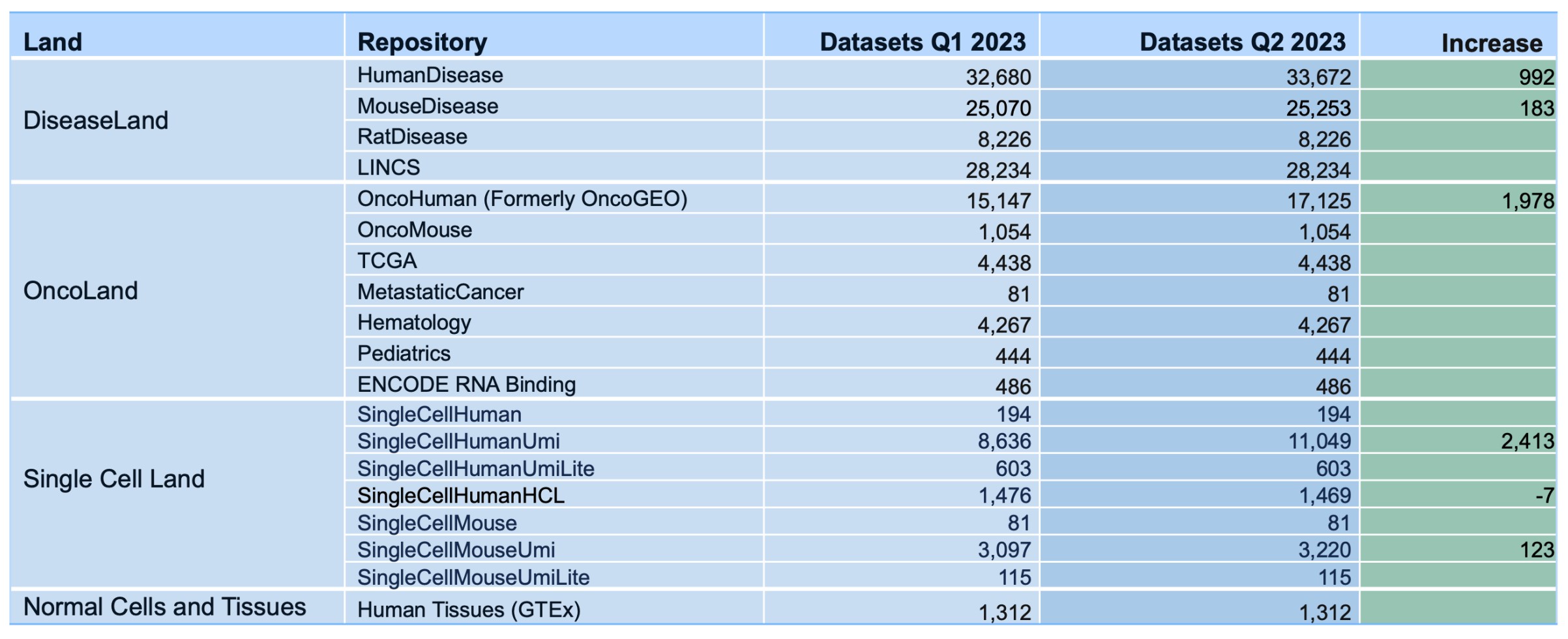
OmicSoft 資料集按 Land 劃分的情況 (到 2023 年 7 月)
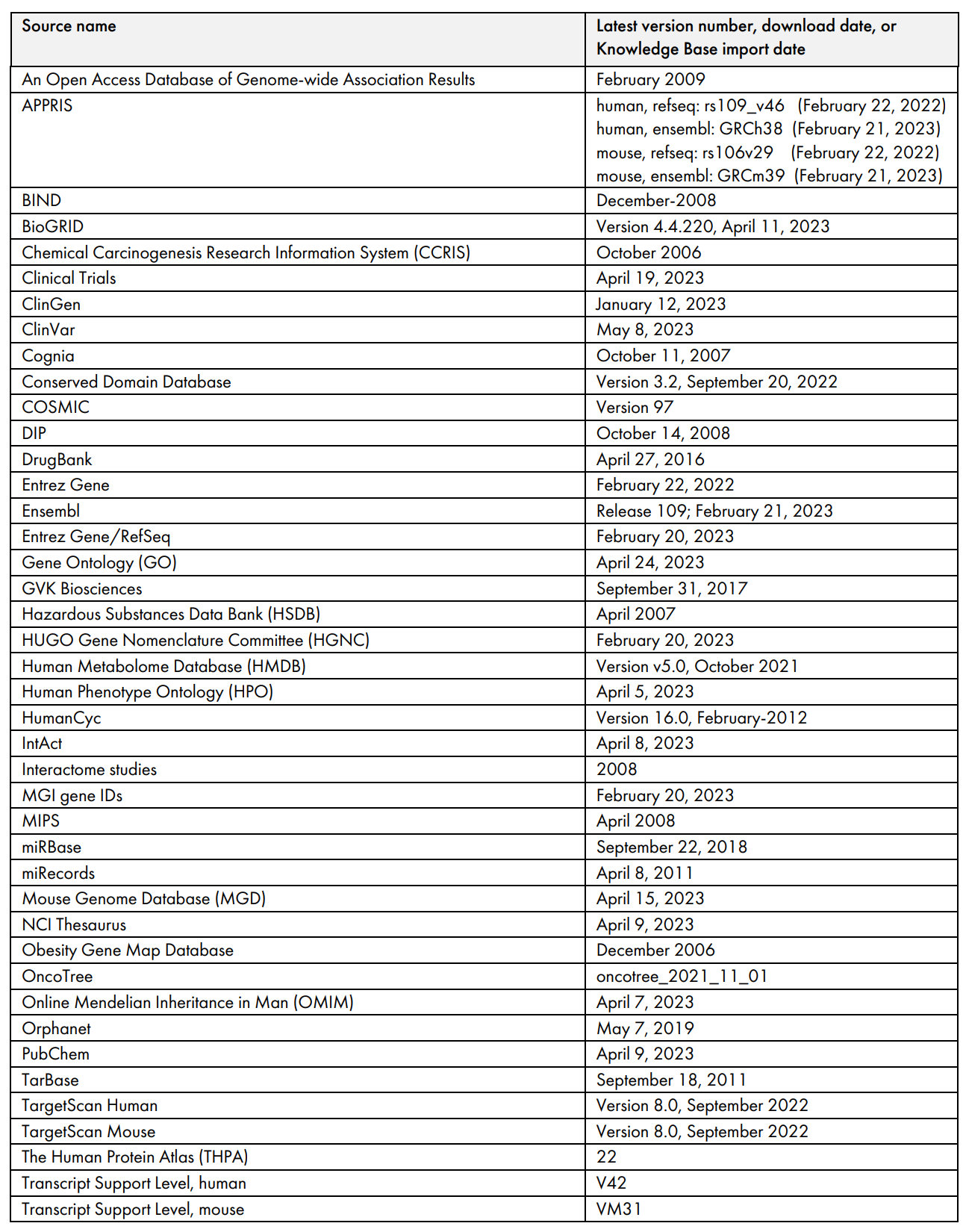
第三方資料庫的版本和/或日期 (到 2023 年 7 月)
若有需要進一步的資訊或在使用軟體上遇到問題歡迎聯繫以下窗口:
席姵妤 業務專員
02-2795 1777 #3013
周儀柔 資深業務專員
02-2795 1777 #3019
黃柔諭 產品專員
02-2795 1777 #3024
訊聯基因數位股份有限公司 (原名:創源生技) | 分子數位中心
台北市 114 內湖區新湖一路 36 巷 28 號