QIAGEN IPA Spring Release (2023 春季更新)
1. 根據基因 網路和路徑辨認潛在的細胞類型
IPA現在可以根據您的網路或路徑上的基因預測相關的細胞類型。根據路徑上的基因組與已知特定細胞類型中表達的基因組數據庫進行富集計算來預測細胞類型。特定細胞類型的基因表達數據來自人類蛋白質地圖 (The Human Protein Atlas) 。
圖 1 顯示了更新的 Cells and Tissues 功能, 範例的基因網路來自自然殺手細胞單細胞群的數據 (人類胎兒肝臟, PMID 31597962)。 如預期的,結果顯示該網路富含自然殺手細胞基因 (p-value 2.04E-20)。
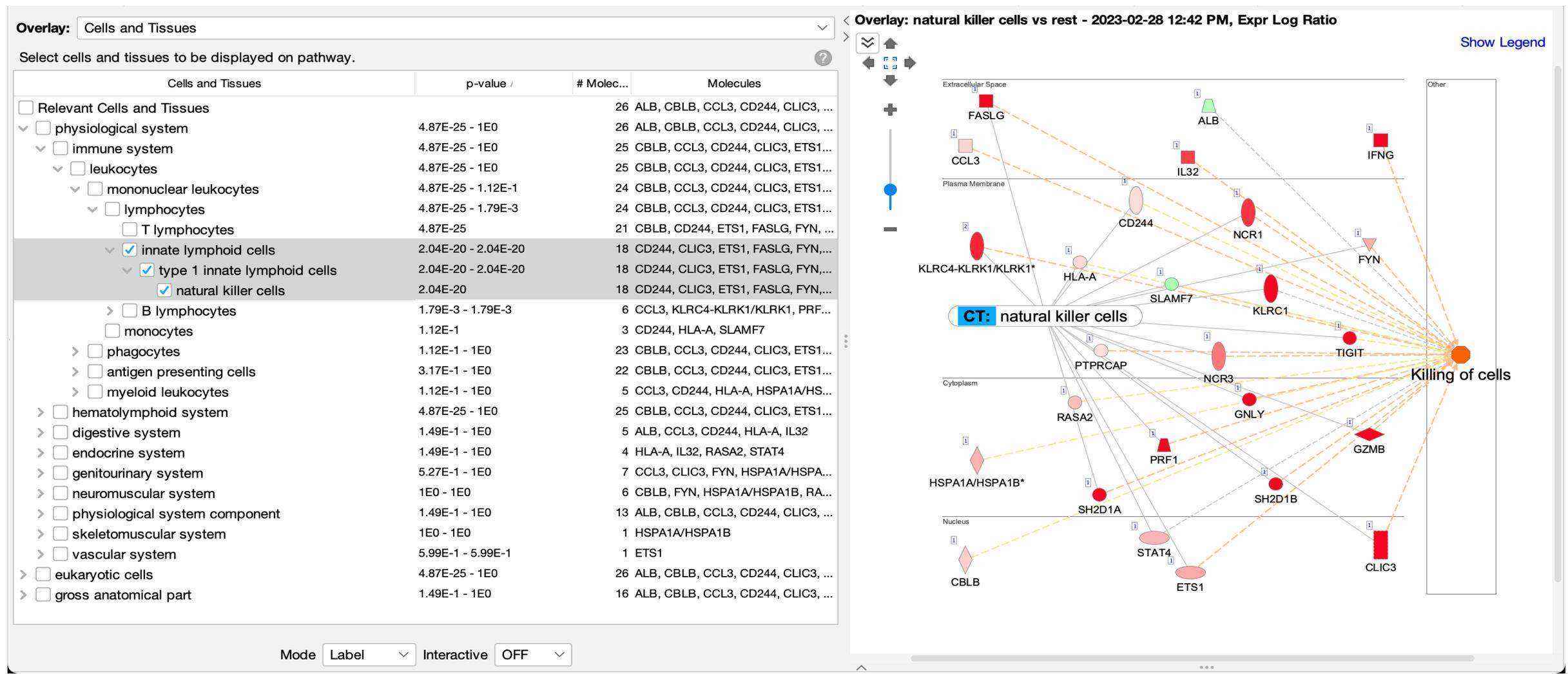
圖 1、 基因網路上富集的自然殺手基因。
在發現基因網路富集自然殺手細胞相關的基因後,標籤 (CT: natural killer cells) 被添加到基因網路上。CT 代表 Cell 或 Tissue 的標籤 。細胞類型判斷的基準被定義為,在一個特定細胞類型中基因表現量是 The Human Protein Atlas中的其他細胞類型表現量的三倍。
這些細胞類型是由 Ingenuity -Ontology 的三個主要分支組成,分別為生理系統 (physiological system), 真核細胞 (eukaryotic cells), 以及解剖 (gross anatomical)。特定的細胞類型通常在這三個主要分支下層 2-3個分支中被發現。在圖1的例子中,自然殺手細胞在免疫系統 (immune system) 底下被發現 (在生理系統 (physiological system) 分支的底下 ),並且如圖2所示,自然殺手細胞也在真核細胞 (eukaryotic cells) 分支下的血球細胞 (blood cells) 中。

圖 2、自然殺手細胞亦屬於 Ingenuity -Ontology 的真核細胞分支。
在這次 IPA 的更新,細胞類型數據來自「 RNA 單細胞類型組織分群 」,其代表79個來自人類蛋白質地圖資料庫的不同細胞類型分群。 IPA 預計在未來的更新中添加更多細胞和組織類型的來源。
2. 在 Core analysis中對 My Pathway進行 z-score 計算
有了 IPA 的這個新功能,您可以在 My Pathway 上設置活化或抑制基因的模式 IPA 便可以藉由該模式比較數據集中分析就緒分子的差異表達來計算 z-score。如此一來,IPA 可以預測在我的數據集中 My Pathway 是被活化或抑制。每個節點的活化狀態(紅色或綠色)可以通過使用 (MAP Molecule Activity Predictor)功能中的紅色或綠色油漆桶進行手動設定,或者通過結合油漆桶和疊加分析進行設定。
圖 3 顯示了在 IPA 中新增 My Pathway 的範例,描述了幾個關鍵的上皮-間質細胞轉換相關基因和生物學功能 。基因節點已用 MAP 油漆桶著色(紅色表示活化 ,綠色表示抑制)。一旦路徑被儲存並用於 z-score 計算,路徑就可以在之後的 Core Analysis 下進行 z-score 計算 。
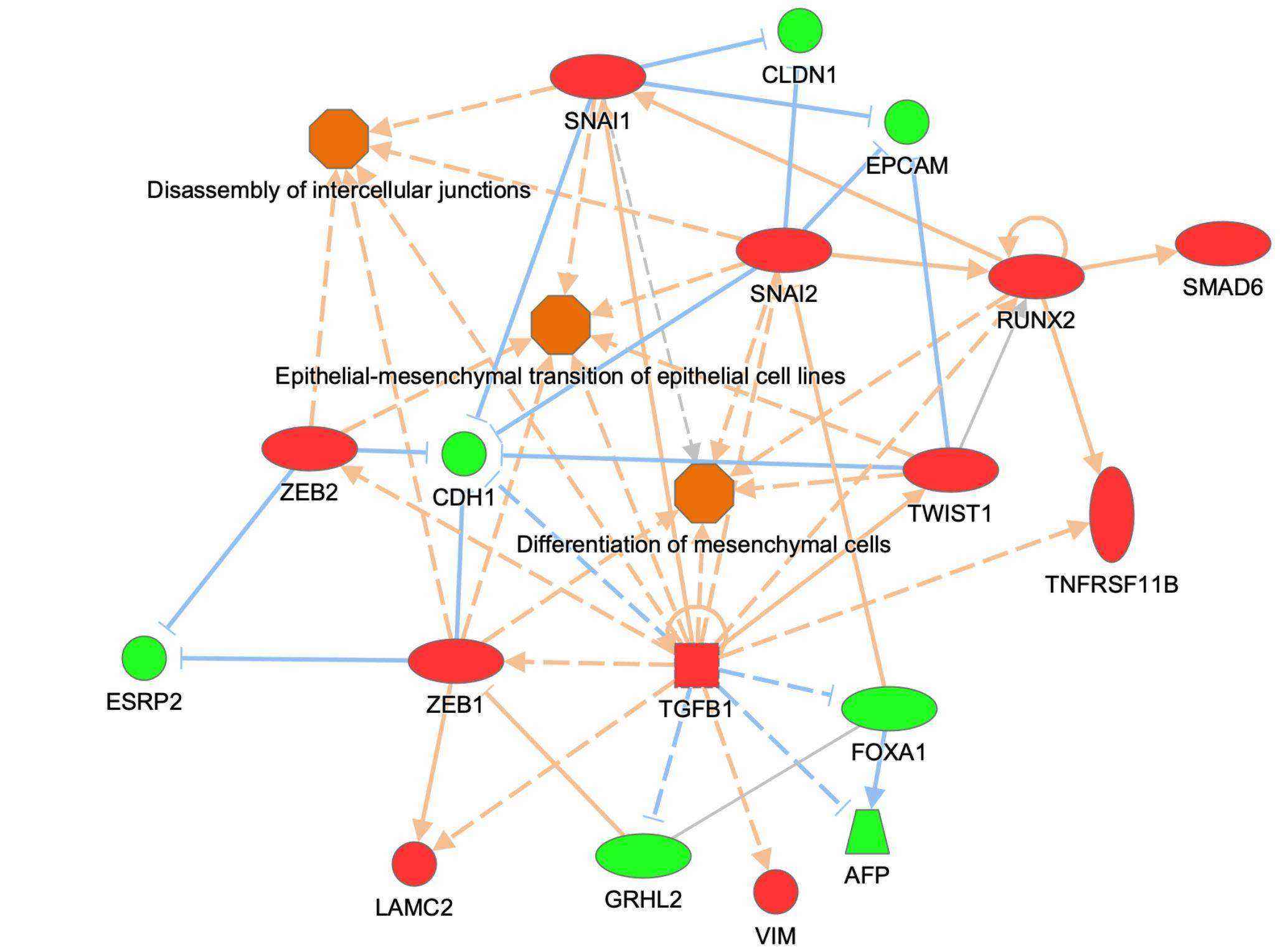
圖 3、 設定 My Pathway,其節點由 使用者設定 為 活化 (紅色)或 抑制 綠色 。
這個路徑可以在之後的 Core Analysis被儲存及計算 z-score。請注意,任何橙色或藍色的分子或疾病並未被保存為路徑的一部分。
分數計算方式是利用 z-score 算法,類似於 Canonical Pathways 的計算方式,方法為將數據中表現量上升或下降的分子和已儲存在 My Pathway 中活化狀態 (紅色或綠色) 配對的分子進行比較。圖 4 顯示了一個來自緊密連接蛋白低表達型乳腺癌細胞株與管腔細胞株比率的基因表達數據 (PMID 20813035) 進行 Core Analysis後 的 My Pathway 分頁 。
IPA 預測上皮細胞間質轉化路徑 (EMT pathway) 在轉移性癌細胞株中被活化,對於該細胞株這是預期的結果。因為數據中實際的表達方向 (圖 4中下方表格的第四列 ) 與 My Pathway 中表現的方向一致 (圖 4 中下方表格的第七列所以 z-score 被計算為正值 。
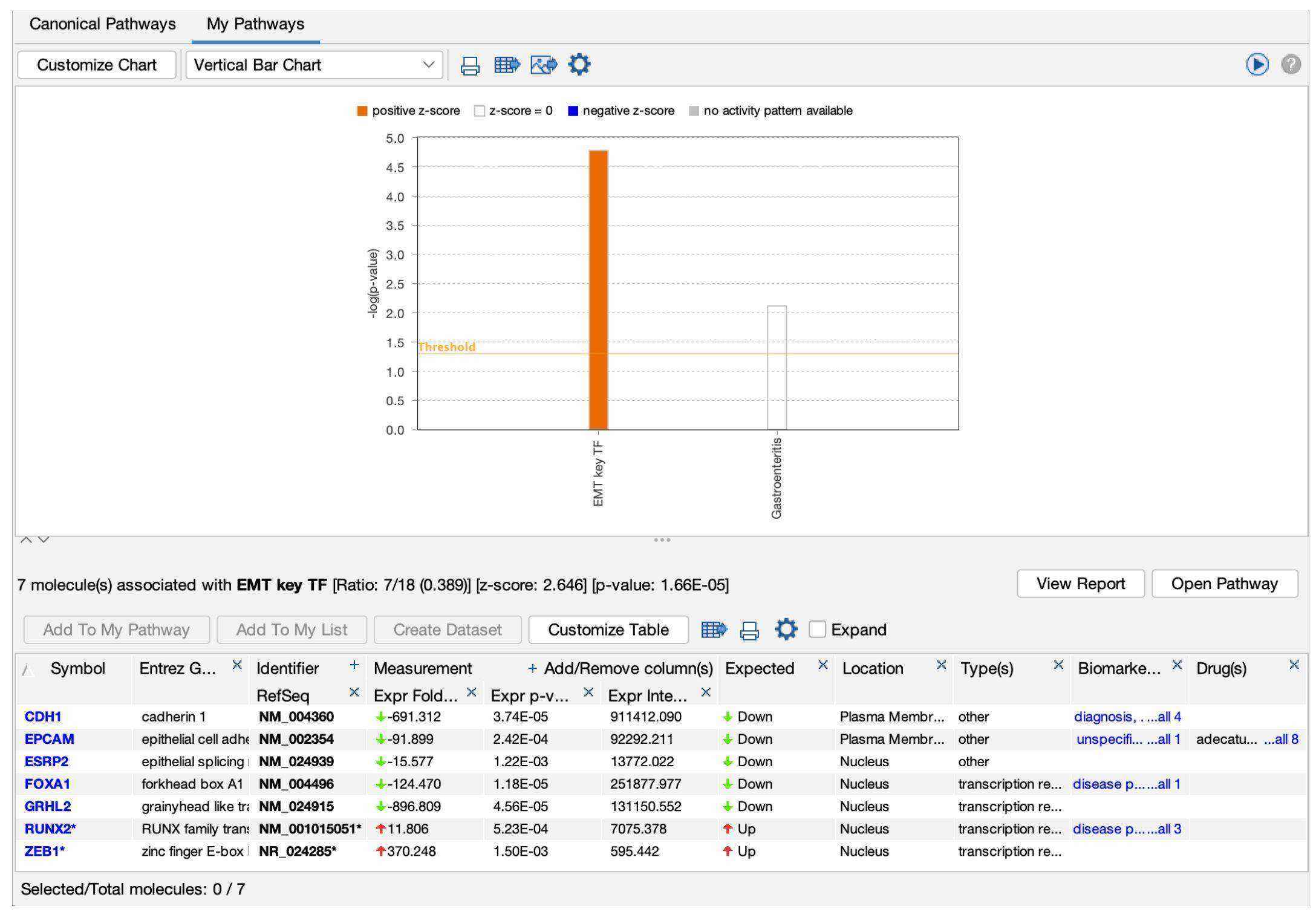
圖 4、 My Pathway 中的因果得分。
在圖 3 中 My Pathway的「 EMT key TF」分數已經在 Core Analysis 中被計算,並且在上方的橙色條表示。橙色表示該路徑在轉移性乳癌細胞株表現量中被預測為活化。如圖表中所示,該路徑的 z-score 為 2.646。
這項新功能提供您建立任何路徑並探討它們在實驗組中的影響。路徑上的基因不需要通過關係連接 。您還可以修改生物路徑或其他 IPA 路徑作為您的「 My Pathway 」的起點 。
3. 上傳數據集時將「 使用者 數據集 」設置為參考數據
在分析數據集時,用於統計計算的基因「 範圍 」定義應盡可能與您在實驗設置中測量(或可以測量)的基因集最接近。例如,如果您正在分析一個包含 400 個基因的面板,那麼這個「 參考數據 」應該是這 400 個基因(最好是在目前的實驗條件下可測量的基因子集 )。如果您的數據只能測量這 400 個基因的變化,卻將參考數據設置為基因組中的所有基因,會在統計上是不正確的。
舉例來說,如果您正在對老鼠腎臟組織進行全轉錄組 RNA-Seq,那麼理想的參考基因數據將是您實驗中可測量到的所有基因,例如在至少一個樣本中通過某些閾值的基因(例如 RPKM>1)。這樣範圍被設定為「 老鼠腎臟表達的基因 」,而不是基因組中所有可能的基因因為其中一些在老鼠腎臟中不被表達。
IPA 允許您上傳可檢測到的所有分子數據,並在分析數據時將「 使用者數據集 」設置為參考數據 。然而,在建立分析時容易忘記使用該設定,導致整個基因組被設定為統計計算的基因範圍 。在此版本的 IPA 中,您可以在上傳數據集時將參考數據設置為「 使用者數據集 」,這樣您更容易記得正確地設置。
圖 5 顯示了新的上傳設定 。這個新功能應該可以減少在分析中意外使用不太理想的參考數據。
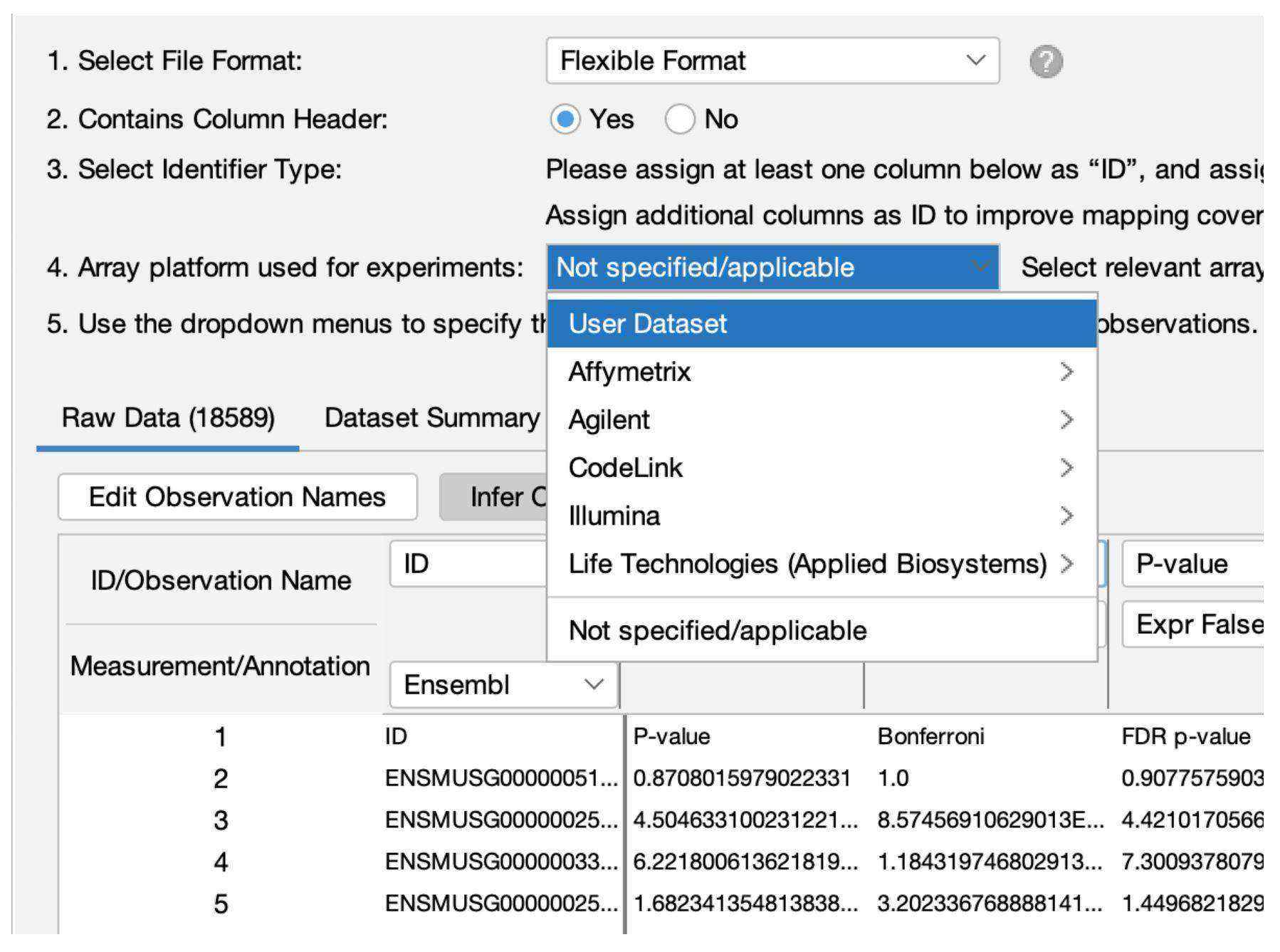
圖5、在數據上傳時將參考數據設置為使用者數據集 (User Dataset)。
請注意,如果您的數據集僅包含您實驗中的差異表達基因,則不應使用「 User Dataset」做為參考數據的選項。在這種情況下,如果您在分析時沒有設置更嚴格的篩選標準,那麼統計結果將是不正確的 。因為在這種情況下,分析就緒的基因和參考數據基因之間沒有區別。統計分析的設計是為了在可能的基因數據範圍中找到一個更小的富集基因組。
4. Graphical Summary 圖例
查看 Graphical Summary Core Analysis 中的分頁時,圖例會出現在屏幕的右上角,如圖 6 所示 。
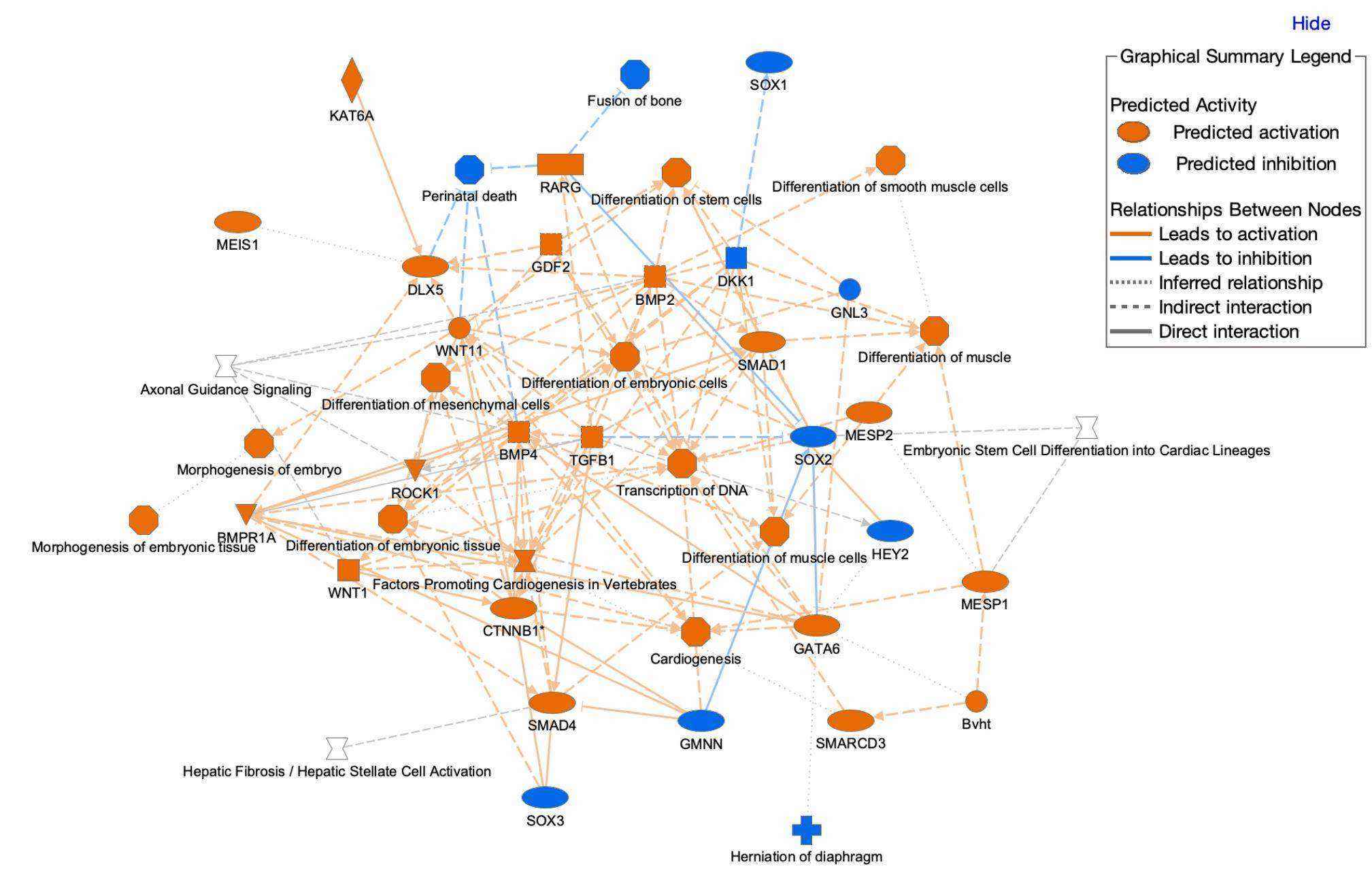
圖 6 Graphical Summary 圖例
圖例顯示在右上角。
5. 資料庫更新
New pathways
- ABRA Signaling Pathway
- Activin Inhibin Signaling Pathway
- DHCR24 Signaling Pathway
- WNK Renal Signaling Pathway
Existing pathways updated to include an activity pattern
- RAR Activation
- Role of Tissue Factor in Cancer
- Serotonin Receptor Signaling
- Transcriptional Regulatory Network in Embryonic Stem Cells
Addition of >263,000 new findings (bringing the total in IPA to over 12.3 million)
>66,000 Expert findings
>24,000 protein-protein interaction findings from BioGrid
>500 protein-protein interaction findings from IntAct
>122,000 cancer mutation findings from ClinVar
>650 Gene -Ontology findings
>800 gene to disease associations from -Online Mendelian Inheritance in Man (OMIM)
>1,800 cancer mutation findings from COSMIC
>350 gene to disease findings from ClinGen
>900 target-to-disease findings from ClinicalTrials.gov
>1,100 drug-to-disease findings from ClinicalTrials.gov
>300 gene to disease or phenotype associations from the Mouse Genome Database (MGD or "Jax”)
>42,500 gene to cell type findings from The Human Protein Atlas (THPA)
135,641 expression datasets will be available in mid-April 2023 (10,714 added)
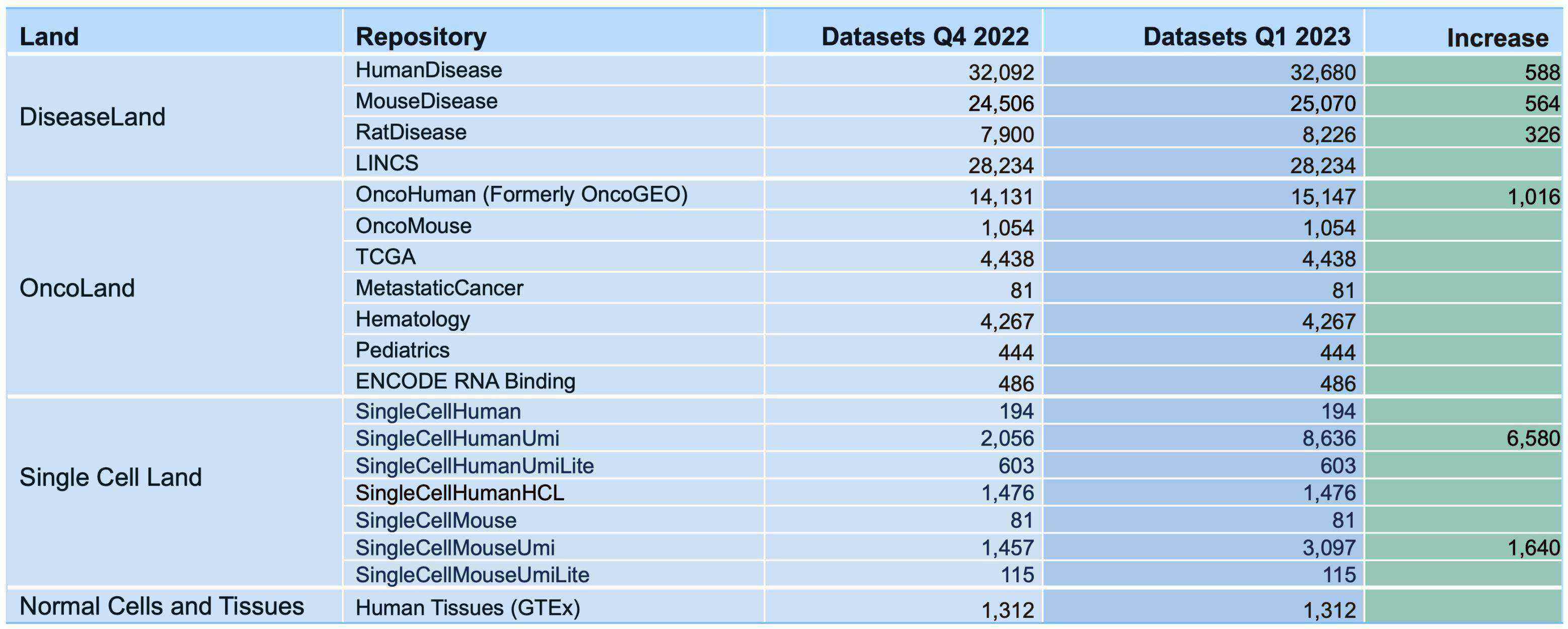
Breakdown of the OmicSoft datasets by Land (mid-April 2023)
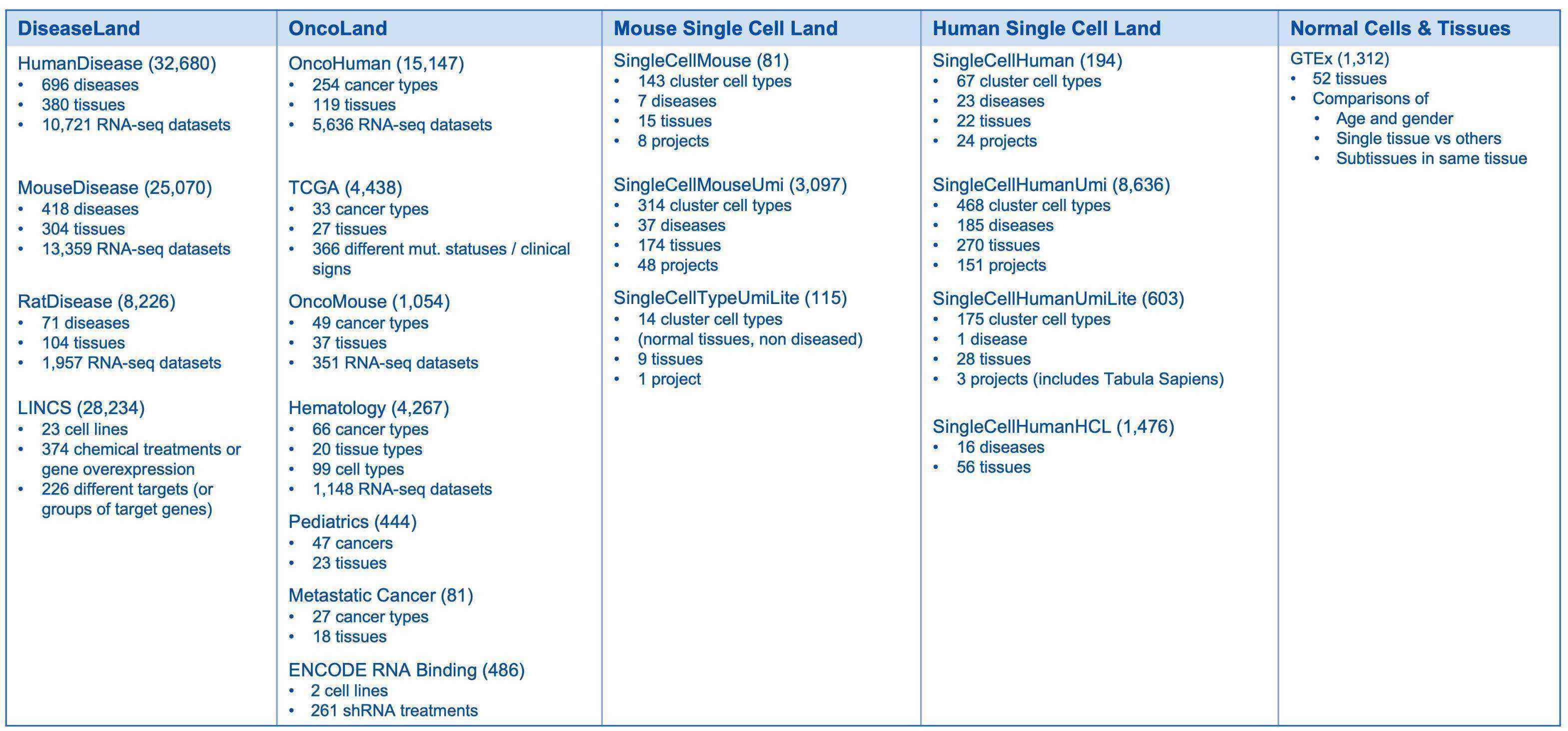
Gene model source versions

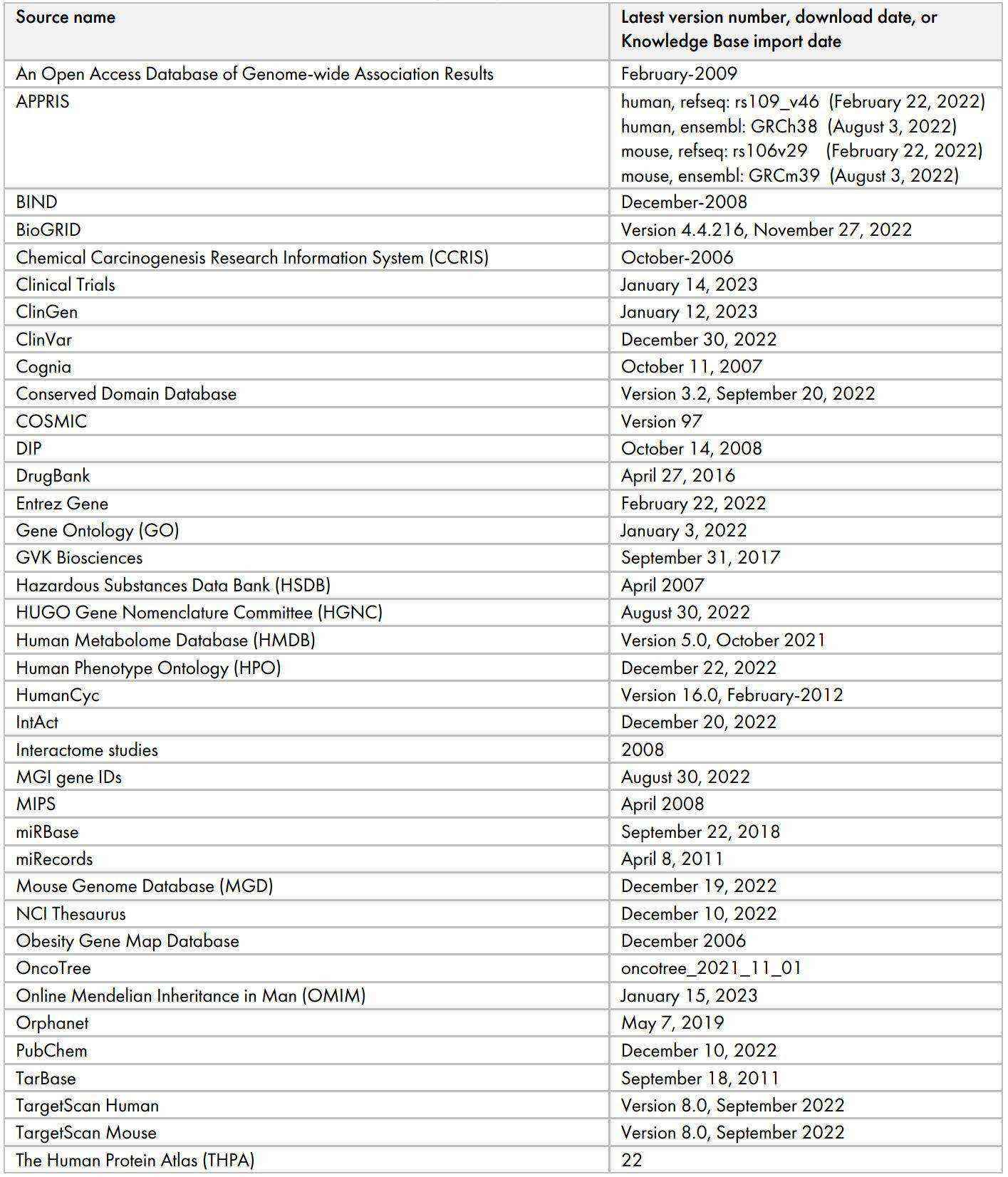
Version and/or date of third-party databases (mid-April 2023)
若有需要進一步的資訊或在使用軟體上遇到問題歡迎聯繫以下窗口 :
周儀柔 資深業務專員 GraceChou@GGA.ASIA 02-2795-1777 #3019
黃柔諭 產品專員 ZoeHuang@GGA.ASIA 02-2795-1777 #3024
訊聯基因數位(原名:創源生技)股份有限公司分子數位中心
台北市 114 內湖區新湖一路 36 巷 28 號

