CLC Genomics Workbench應用於粒線體基因組單倍型分析和系統發育地理學研究
CLC Genomics Workbench應用於粒線體基因組單倍型分析和系統發育地理學研究
人粒線體基因組大小約為16.6 kb,編碼37個基因,包括13個編碼蛋白的基因、22個tRNA和2個rRNA。非編碼區又稱控制區(control region,CR),主要調控粒線體複製和轉錄。該區域包含3個高變區(hypervariable regions,HV-I、HV-II和HV-III),其變異率高於編碼區。因此,其多型性廣泛用於母系遺傳示蹤、人群研究和法醫鑑定。
目前已有多種分析工具能夠解析粒線體單倍群資訊,但通常需要結合多個基於網頁的分析工具。一方面不利於資料安全,另一方面無法推斷地理起源,這些限制了粒線體基因組序列分析和應用。為了解決這些問題,美國德克薩斯大學和布魯克陸軍醫療中心的研究人員基於CLC Genomics Workbench開發了一套本地自動化的分析方案,能夠快速精準地完成序列比對和突變分析,獲得單倍群、地理來源和種族資訊。
粒線體基因組資料庫建構
首先作者建構了hMITO粒線體基因組資料庫,該資料庫由4,286條粒線體基因組序列構成。通過整合文獻中的樣本資訊,該資料庫包含了序列、單倍群、地理來源、突變等資訊。
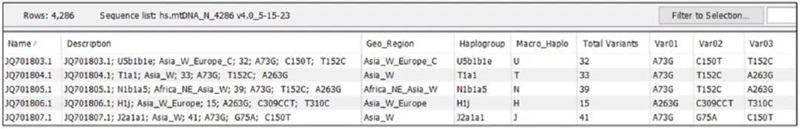
通過CLC Genomics Workbench建構粒線體基因組的進化樹,並新增單倍群和地理資訊,有根樹表明尼安德塔人與現代人的分歧點發生在大約80萬年前。
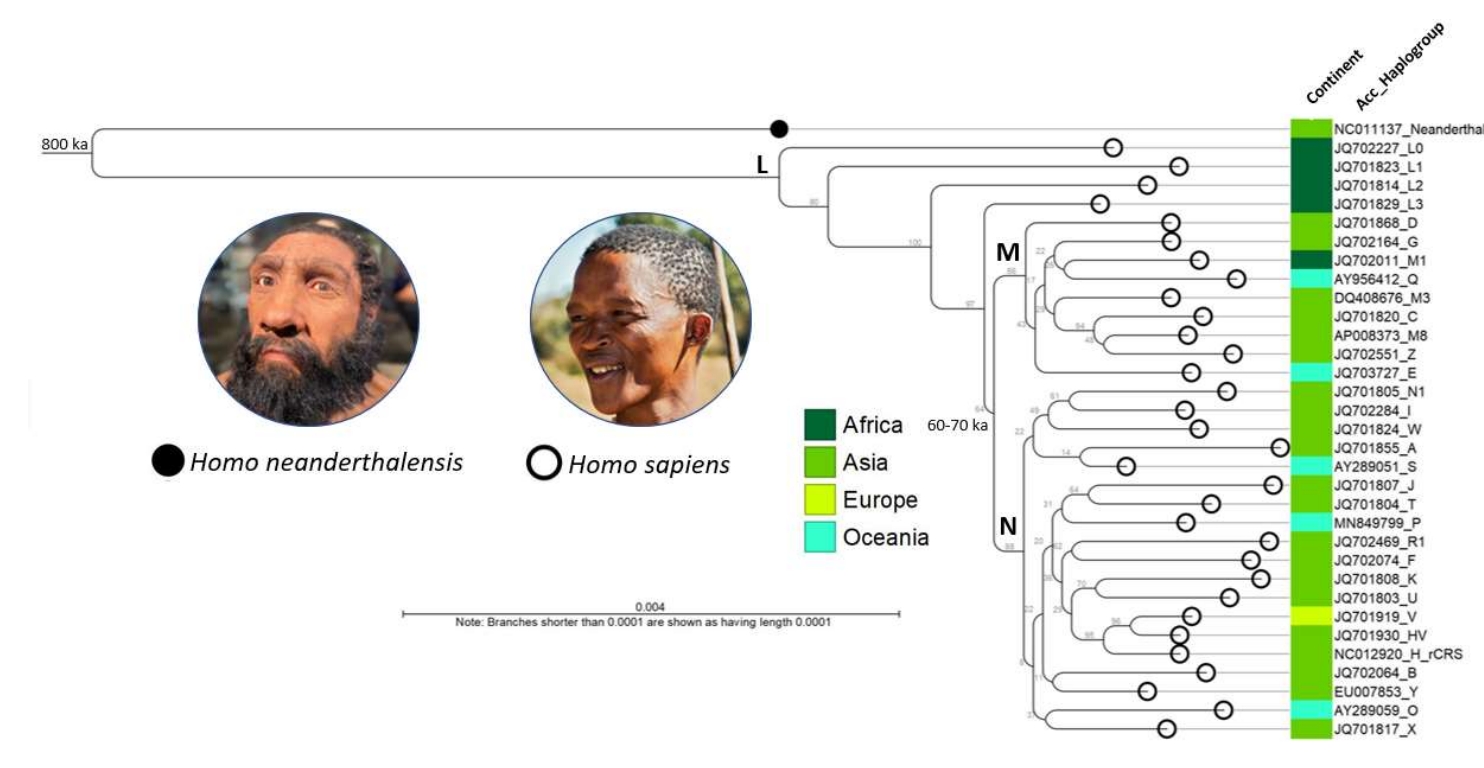
粒線體基因組定序
QIAseq Human Mitochondria Panel可以輕鬆實現粒線體全基因組的擴增及定序。QIAseq Panel藉助分子條形碼和單端特異性引物延伸技術,避免了PCR duplicates、檢測結果假陽性和文庫偏倚等問題,使得檢測結果更加精準可靠,特殊的擴增方式也更加適合降解DNA的檢測。
粒線體基因組資料分析
作者基於CLC靈活的分析流程,建構了一套包含QC、Trim、Mapping、Call variant、De novo assembly和BLAST的一鍵式分析流程。
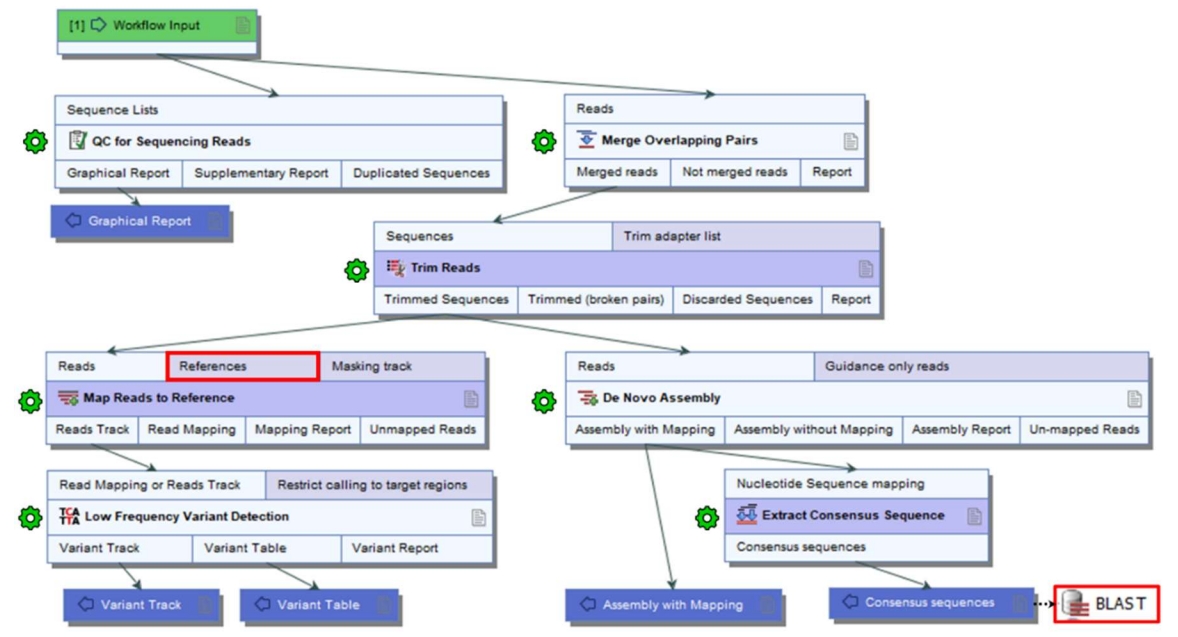
運行該分析流程,可以獲得樣本的突變位點以及該位點的詳細資訊,包括鹼基座標、鹼基突變類型、定序深度、突變頻率等。
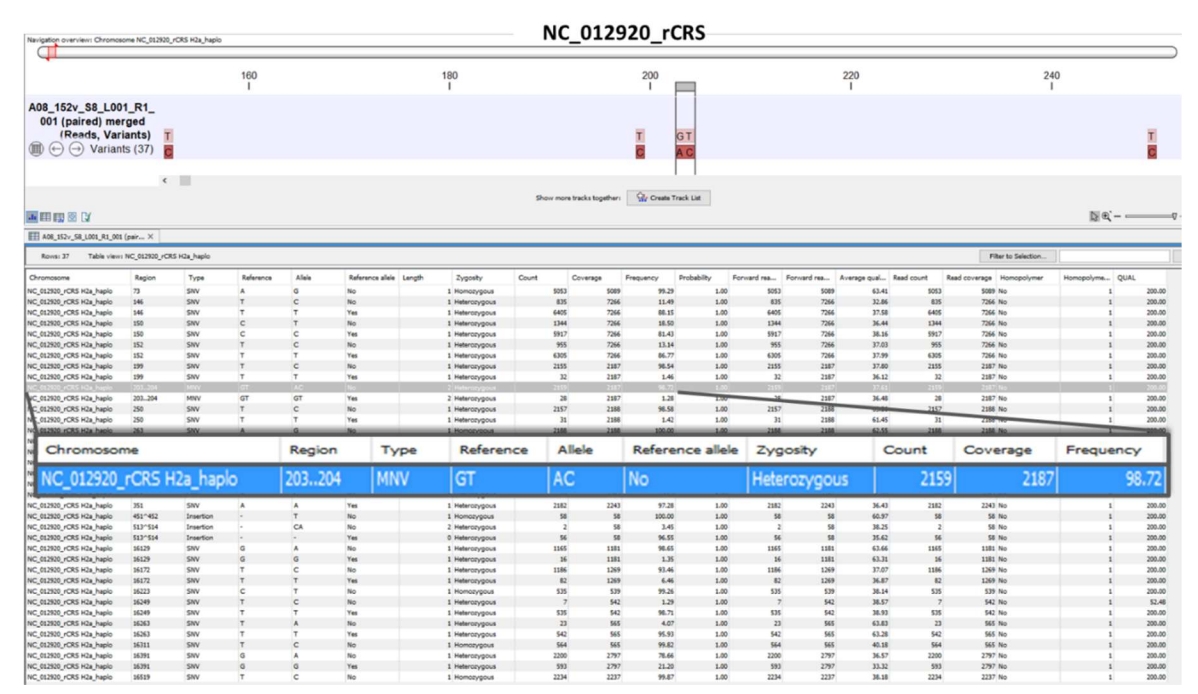
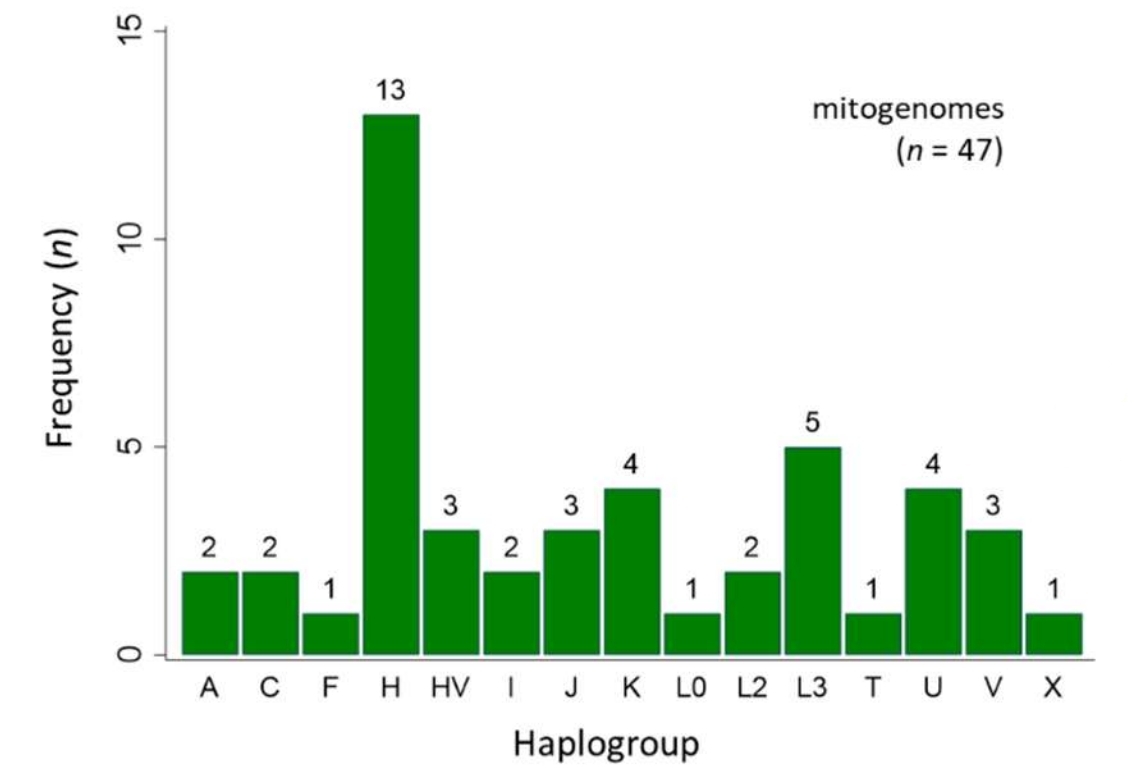
單倍群和種族分析
在得到粒線體基因組一致性序列後,通過BLAST與建構的hMITO粒線體基因組本地資料庫進行比對,從而獲得了所有樣本的單倍群和種族資訊。
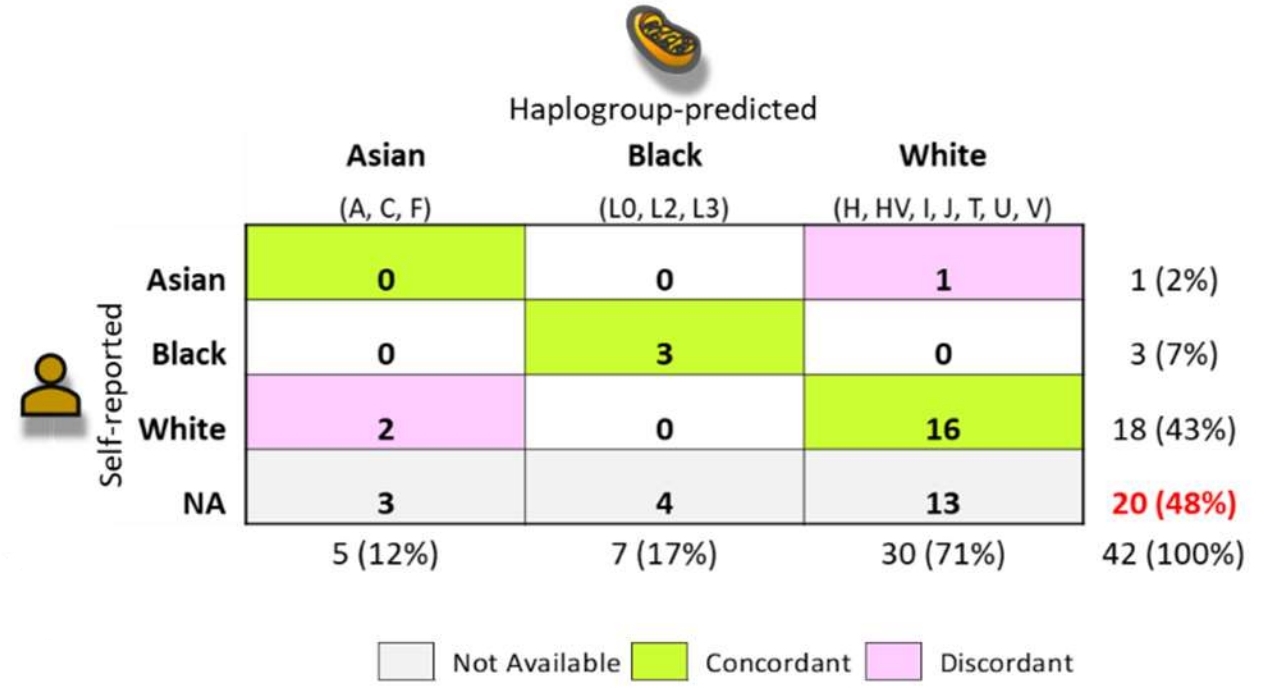
通過與自報告的種族資訊對比,除去20個未報告種族資訊的樣本,在剩下的22個樣本中,19個樣本預測的種族資訊與自報告一致,正確率超過86%。僅有3個樣本預測的種族資訊與自報告不符,主要集中在亞洲人和白人種族。
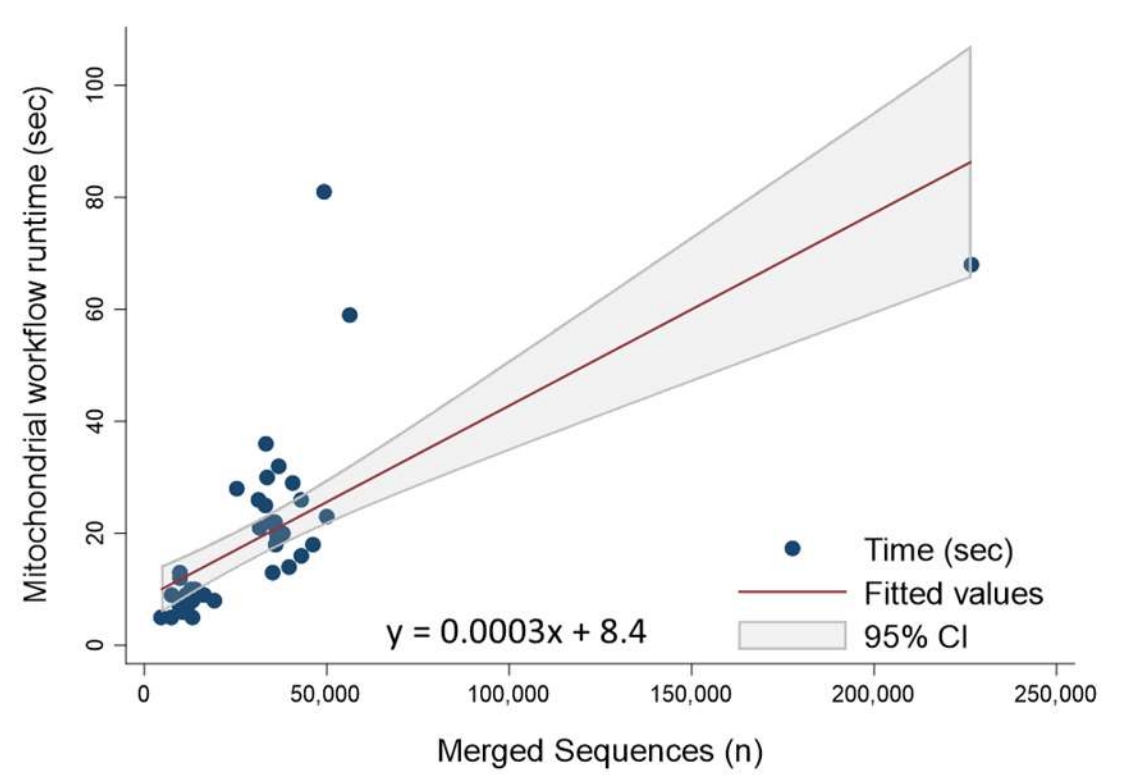
執行階段間
每個樣本包含的合併reads數量從4,580到226,664不等,在Windows10作業系統、雙核Intel i7-7500U處理器、8GB記憶體的電腦上,CLC Genomics Workbench僅需 14.4分鐘即可完成資料分析,分析單個樣本的中位數時間僅為13秒。
綜上所述,CLC Genomics Workbench能夠本地化快速完成人粒線體基因組資料分析,獲得單倍群、地理來源和種族資訊。CLC的強大功能不僅於此,它還可以分析轉錄組資料、單細胞資料、宏碁因組資料等。
參考文獻
https://doi.org/10.20944/preprints202307.1891.v1

